Compara métodos de campo y dron (paper SUDOE)
ajpelu
2022-04-18
Last updated: 2022-04-20
Checks: 7 0
Knit directory:
veg_alcontar/
This reproducible R Markdown analysis was created with workflowr (version 1.7.0). The Checks tab describes the reproducibility checks that were applied when the results were created. The Past versions tab lists the development history.
Great! Since the R Markdown file has been committed to the Git repository, you know the exact version of the code that produced these results.
Great job! The global environment was empty. Objects defined in the global environment can affect the analysis in your R Markdown file in unknown ways. For reproduciblity it’s best to always run the code in an empty environment.
The command set.seed(20211007) was run prior to running the code in the R Markdown file.
Setting a seed ensures that any results that rely on randomness, e.g.
subsampling or permutations, are reproducible.
Great job! Recording the operating system, R version, and package versions is critical for reproducibility.
Nice! There were no cached chunks for this analysis, so you can be confident that you successfully produced the results during this run.
Great job! Using relative paths to the files within your workflowr project makes it easier to run your code on other machines.
Great! You are using Git for version control. Tracking code development and connecting the code version to the results is critical for reproducibility.
The results in this page were generated with repository version 95151d5. See the Past versions tab to see a history of the changes made to the R Markdown and HTML files.
Note that you need to be careful to ensure that all relevant files for the
analysis have been committed to Git prior to generating the results (you can
use wflow_publish or wflow_git_commit). workflowr only
checks the R Markdown file, but you know if there are other scripts or data
files that it depends on. Below is the status of the Git repository when the
results were generated:
Ignored files:
Ignored: .Rhistory
Ignored: .Rproj.user/
Ignored: analysis/.DS_Store
Untracked files:
Untracked: analysis/compara_methods_SUDOE_files/
Note that any generated files, e.g. HTML, png, CSS, etc., are not included in this status report because it is ok for generated content to have uncommitted changes.
These are the previous versions of the repository in which changes were made
to the R Markdown (analysis/compara_methods_SUDOE.Rmd) and HTML (docs/compara_methods_SUDOE.html)
files. If you’ve configured a remote Git repository (see
?wflow_git_remote), click on the hyperlinks in the table below to
view the files as they were in that past version.
| File | Version | Author | Date | Message |
|---|---|---|---|---|
| Rmd | 95151d5 | ajpelu | 2022-04-20 | include new analysis |
| html | 968eb41 | ajpelu | 2022-04-19 | Build site. |
| Rmd | 4104f26 | ajpelu | 2022-04-19 | add new correlations |
| html | 8f01211 | ajpelu | 2022-04-19 | Build site. |
| Rmd | 1b5a50d | ajpelu | 2022-04-19 | add analysis diversidad |
| html | 3648c33 | ajpelu | 2022-04-19 | Build site. |
| Rmd | a5285f3 | ajpelu | 2022-04-19 | add analysis diversidad |
| html | bf70f7c | ajpelu | 2022-04-19 | Build site. |
| Rmd | 586d4b9 | ajpelu | 2022-04-19 | add new analysis organized |
1 Objetivo 1. Carga de combustible: cobertura y fitovolumen
1.1 Cobetura
Vamos a realizar la comparación seleccionando para cada parcela (n=12) un valor de cobertura de quadrats (quadrats medio).
1.1.1 Summary values
| metodo | mean | sd | se | cv | median | min | max | n |
|---|---|---|---|---|---|---|---|---|
| line_intercept | 27.19 | 6.49 | 1.87 | 23.86 | 29.67 | 11.95 | 34.0 | 12 |
| point_quadrat | 55.50 | 7.62 | 2.20 | 13.73 | 56.00 | 41.00 | 64.0 | 12 |
| quadrat medio | 29.92 | 5.35 | 1.54 | 17.88 | 30.81 | 21.25 | 37.5 | 12 |
1.1.2 Comparación de métodos
- ANOVA Kruskal Wallis
| statistic | p.value | parameter | method | mi_variable |
|---|---|---|---|---|
| 23.84398 | 6.6e-06 | 2 | Kruskal-Wallis rank sum test | cobertura |
Los resultados de la ANOVA no paramétrica (Kruskal-Wallis) indican que existen diferencias significativas para la cobertura (%) entre los diferentes métodos de campo empleados (\(\chi^2\) = 23.84; p<0.0001) (Tabla 1.2).
Posteriormente, evaluamos si existen diferencias entre cada uno de los métodos (post hoc) y observamos que existen diferencias significativas entre el point quadrat y los otros métodos (line intercept y quadrat medio) (Tabla 1.3, Figura 1.1). Asimismo, no observamos diferencias entre la cobertura estimada según el line intercept y el quadrat medio.
| H0 | statistic | p.value |
|---|---|---|
| line_intercept = point_quadrat | 4.53 | <0.001 |
| line_intercept = quadrat medio | 0.70 | >0.999 |
| point_quadrat = quadrat medio | 3.84 | <0.001 |
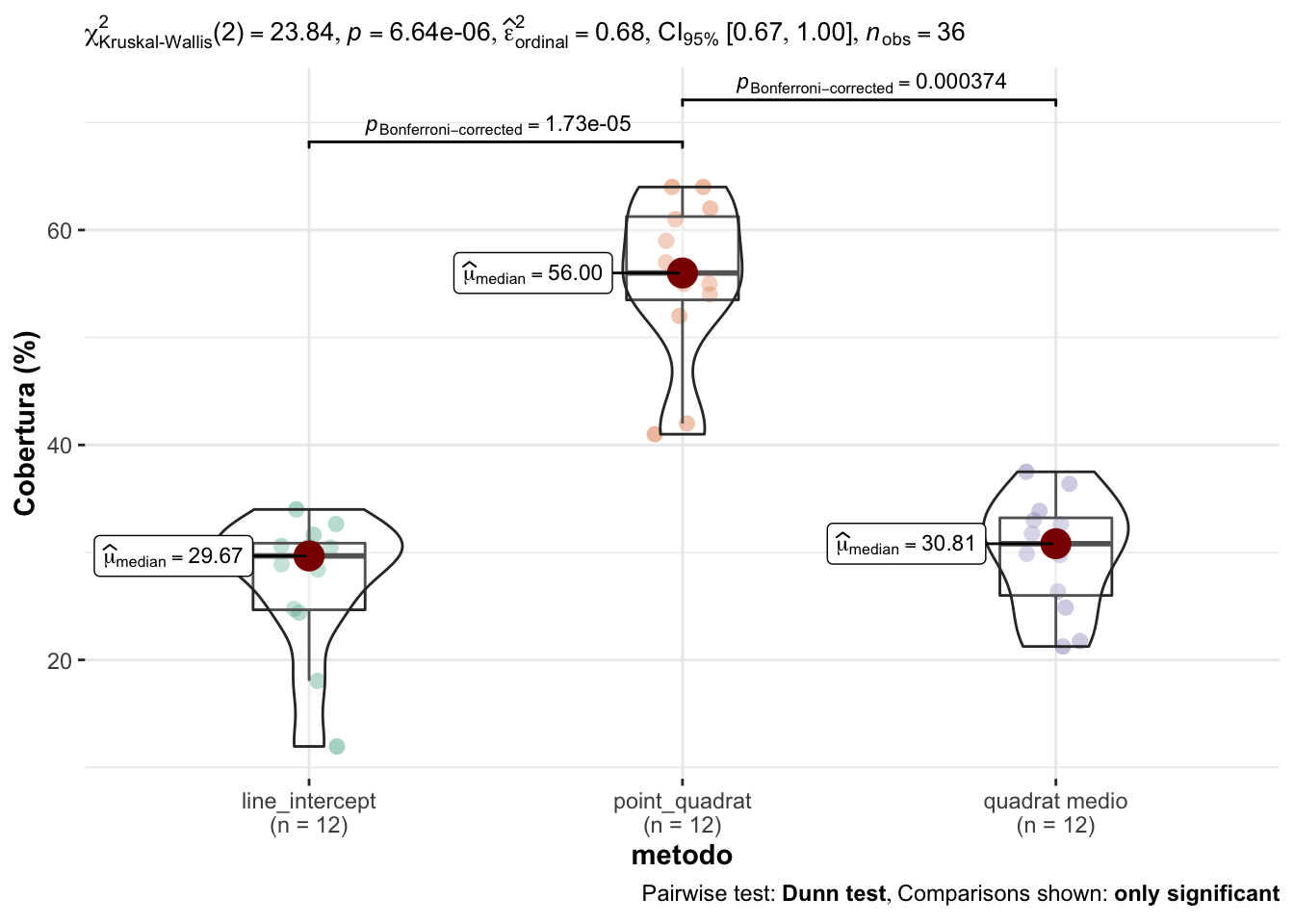
Figure 1.1: Comparación de los valores de cobertura entre los diferentes métodos de campo.
| Version | Author | Date |
|---|---|---|
| bf70f7c | ajpelu | 2022-04-19 |
1.1.3 Correlación
El siguiente paso es evaluar la correlación que existe entre los métodos de campo para la cobertura.
Tal y como observamos en la Figura 1.2, existe una correlación significativa del line intercept con el point quadrat (\(R^2=\) 0.63), aunque lejos del ajuste perfecto (línea negra en Figura 1.2). El método point quadrat sobreestima los valores de cobertura con respecto al método de line intercept. Así, el rango de cobertura estimado por el LI varía entre 11.95-34 %, mientras que la estimación por PQ varía entre 41-64% (Tabla 1.1). La correlación entre quadrat medio y point quadrat es muy baja (Figura 1.3).
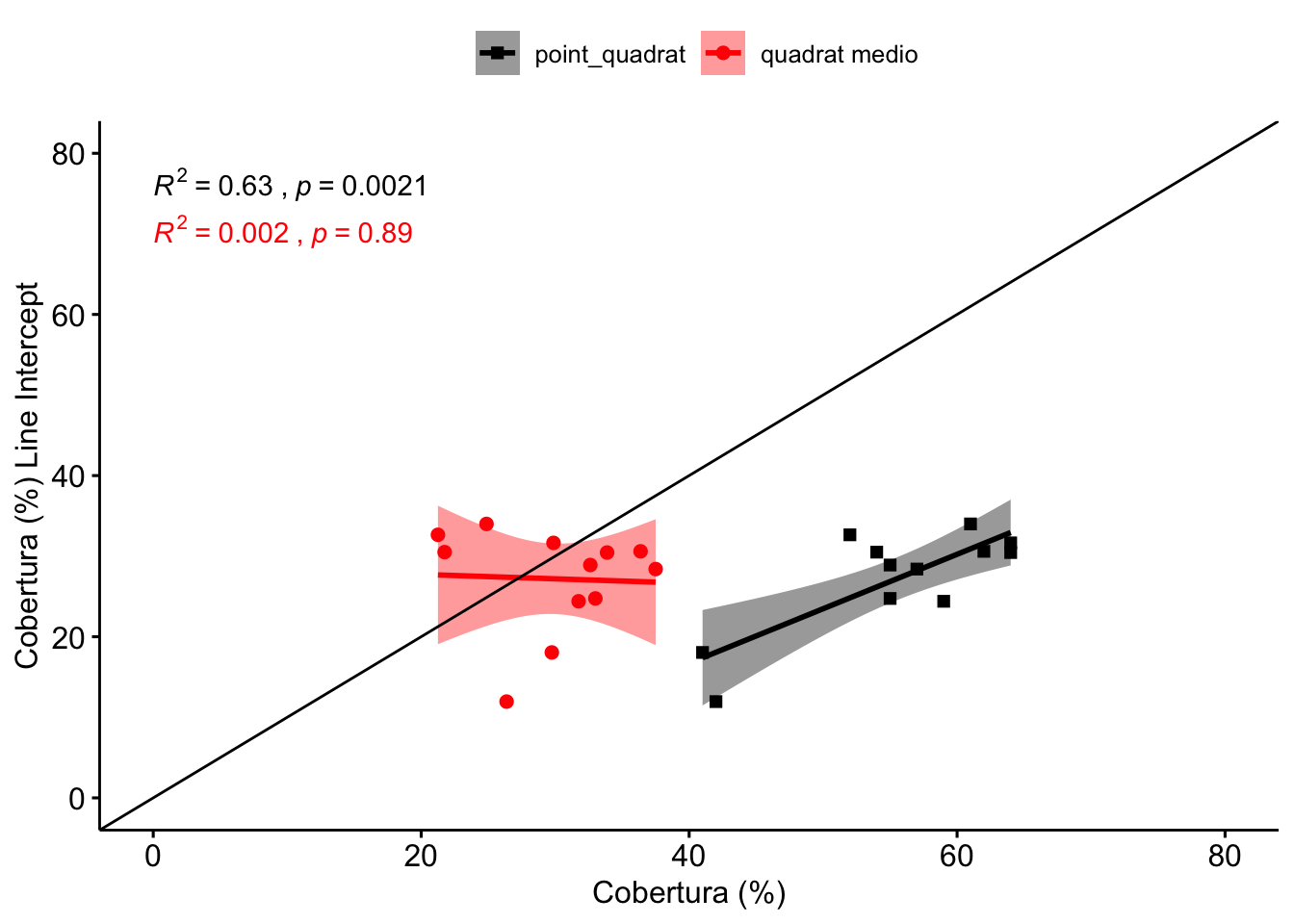
Figure 1.2: Correlación entre los valores de cobertura estimados por Line Intercept y los otros métodos de campo: Point Quadrat y Quadrat medio.
| Version | Author | Date |
|---|---|---|
| bf70f7c | ajpelu | 2022-04-19 |
1.1.3.1 Correlación Quadrat medio - Point Quadrat
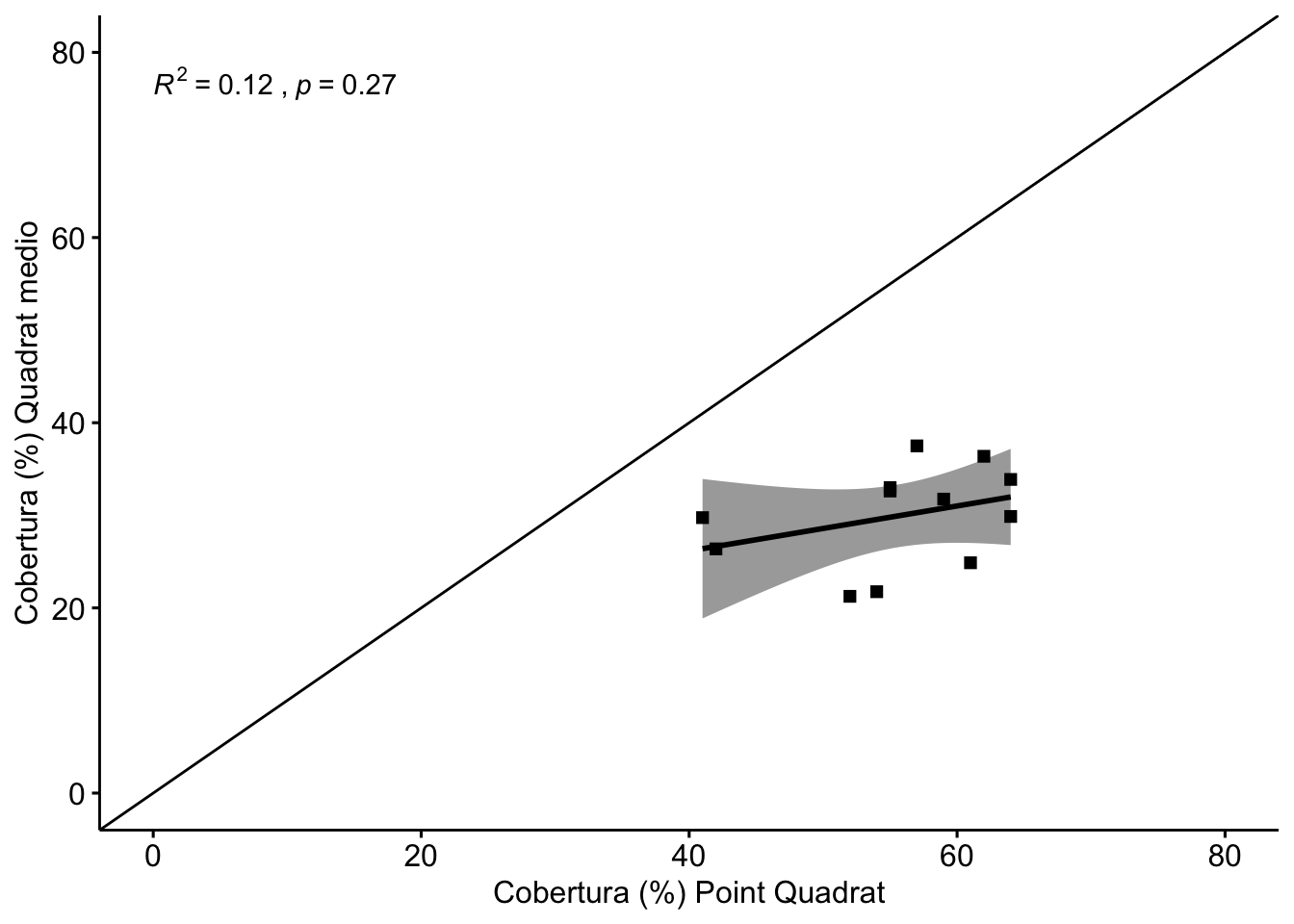
Figure 1.3: Correlación entre los valores de cobertura estimados por Quadrat medio y Point Quadrat.
1.2 Fitovolumen
- En este caso solo compararemos los métodos de Line Intercept y Quadrat medio
1.2.1 Summary values
| metodo | mean | sd | se | cv | median | min | max | n |
|---|---|---|---|---|---|---|---|---|
| line_intercept | 531.04 | 274.90 | 79.36 | 51.77 | 543.13 | 84.27 | 1016.86 | 12 |
| quadrat medio | 778.57 | 275.23 | 79.45 | 35.35 | 699.70 | 324.04 | 1296.47 | 12 |
1.2.2 Comparación de métodos
Hemos comprobado Normalidad (W = 0.97; p=0.6350); y homocedasticidad (Bartlett’s K-squared = 0; p=0.9969) y se puede aplicar un método paramétrico, en este caso, la t-student de comparación de medias. Observamos que existen diferencias (Figura 1.4):
The Welch Two Sample t-test testing the difference of value by metodo (mean in group line_intercept = 531.04, mean in group quadrat medio = 778.57) suggests that the effect is negative, statistically significant, and large (difference = -247.53, 95% CI [-480.42, -14.64], t(22.00) = -2.20, p = 0.038; Cohen's d = -0.94, 95% CI [-1.81, -0.05])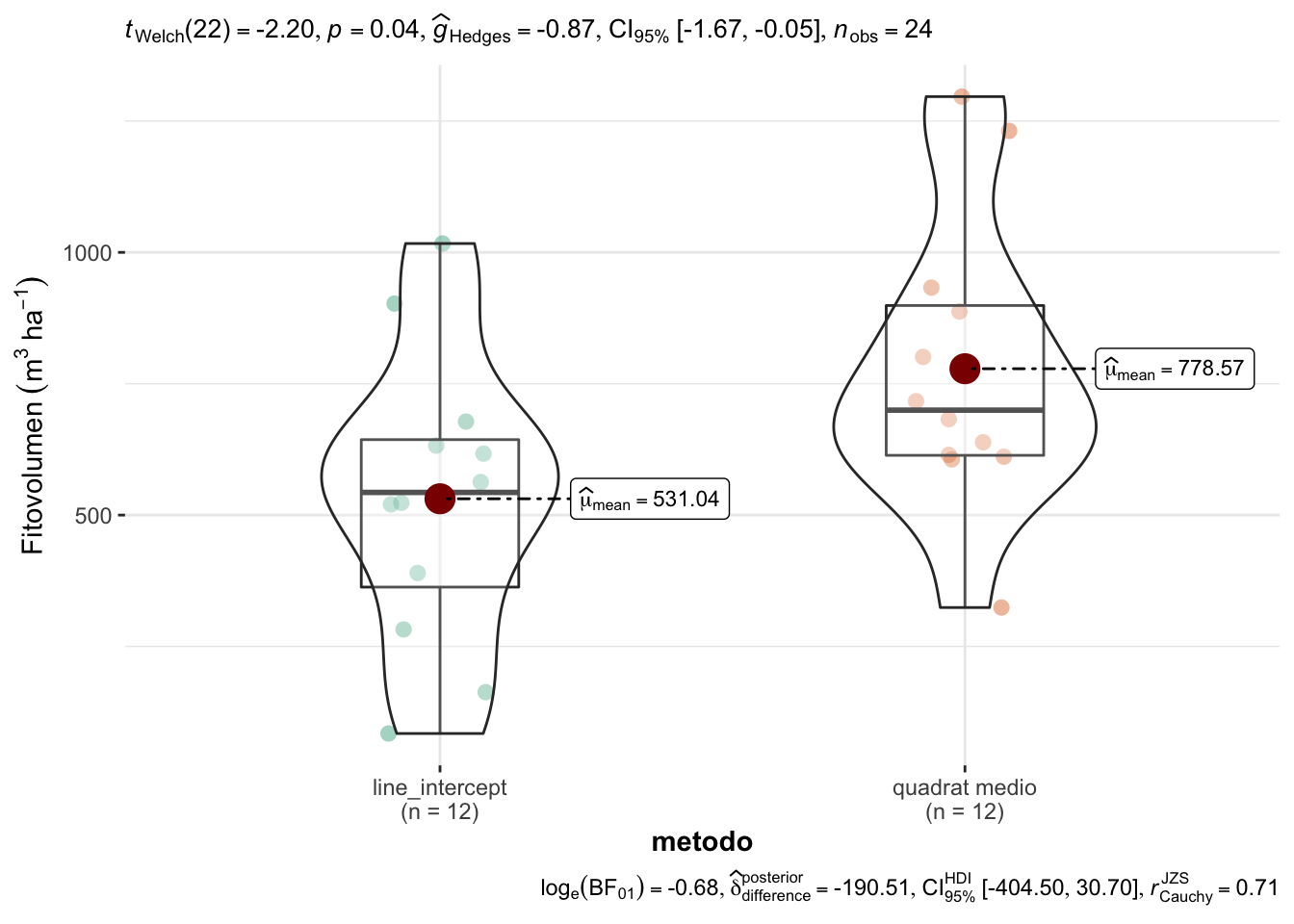
Figure 1.4: Comparación de los valores de fitovolumen entre Line Intercept y Quadrat medio.
| Version | Author | Date |
|---|---|---|
| bf70f7c | ajpelu | 2022-04-19 |
1.2.3 Correlación
No existe una buena correlación entre los valores de fitovolumen estimados con line intercept y los estimados con quadrat medio (Figura 1.5). Este último método sobreestima el fitovolumen registrado por el LI (ver Tabla 1.4).
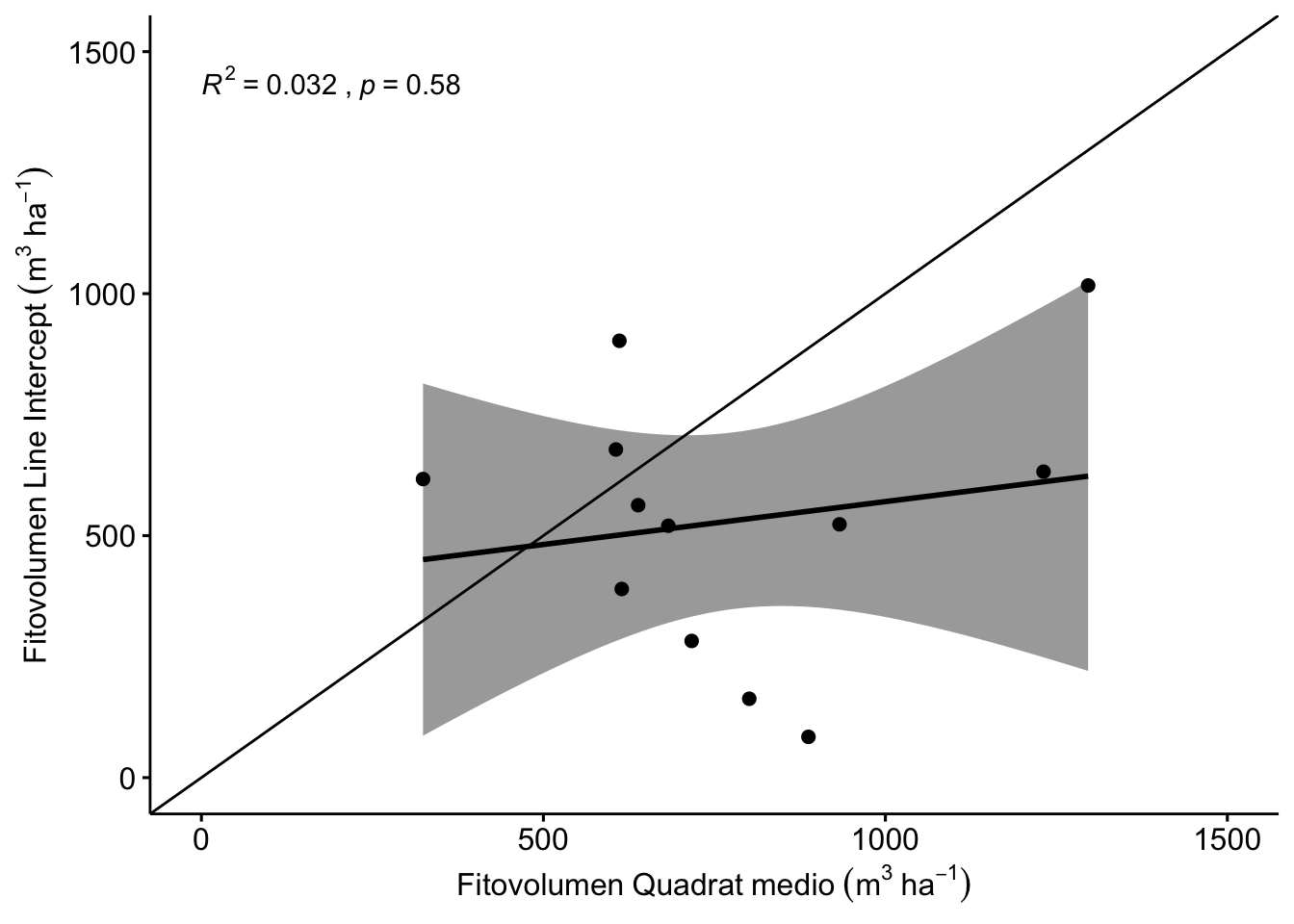
Figure 1.5: Correlación entre los valores de fitovolumen estimados por Line Intercept y Quadrat medio.
| Version | Author | Date |
|---|---|---|
| bf70f7c | ajpelu | 2022-04-19 |
2 Objetivo 2. Diversidad y Riqueza
2.1 Riqueza
Comparamos entre line intercept, quadrat parcela y point quadrat.
2.1.1 Summary values
| metodo | mean | sd | se | cv | median | min | max | n |
|---|---|---|---|---|---|---|---|---|
| line_intercept | 13.00 | 3.10 | 0.90 | 23.88 | 13.0 | 8 | 18 | 12 |
| point_quadrat | 13.17 | 4.24 | 1.22 | 32.20 | 14.0 | 7 | 20 | 12 |
| quadrat_parcela | 34.08 | 10.39 | 3.00 | 30.48 | 34.5 | 19 | 52 | 12 |
2.1.2 Comparación de métodos
- ANOVA Kruskal Wallis
| statistic | p.value | parameter | method | mi_variable |
|---|---|---|---|---|
| 22.86057 | 1.09e-05 | 2 | Kruskal-Wallis rank sum test | riqueza |
Los resultados de la ANOVA no paramétrica (Kruskal-Wallis) indican que existen diferencias significativas para la riqueza de especies vegetales entre los diferentes métodos de campo empleados (\(\chi^2\) = 22.86; p<0.0001) (Tabla 2.2).
Posteriormente, evaluamos si existen diferencias entre cada uno de los métodos (post hoc) y observamos que existen diferencias significativas entre el quadrat parcela y los otros métodos (line intercept y point quadrat) (Tabla 2.3, Figura 2.1). Asimismo, no observamos diferencias entre la riqueza estimada según el line intercept y el point quadrat.
| H0 | statistic | p.value |
|---|---|---|
| line_intercept = point_quadrat | 0.02 | >0.999 |
| line_intercept = quadrat_parcela | 4.15 | <0.001 |
| point_quadrat = quadrat_parcela | 4.13 | <0.001 |
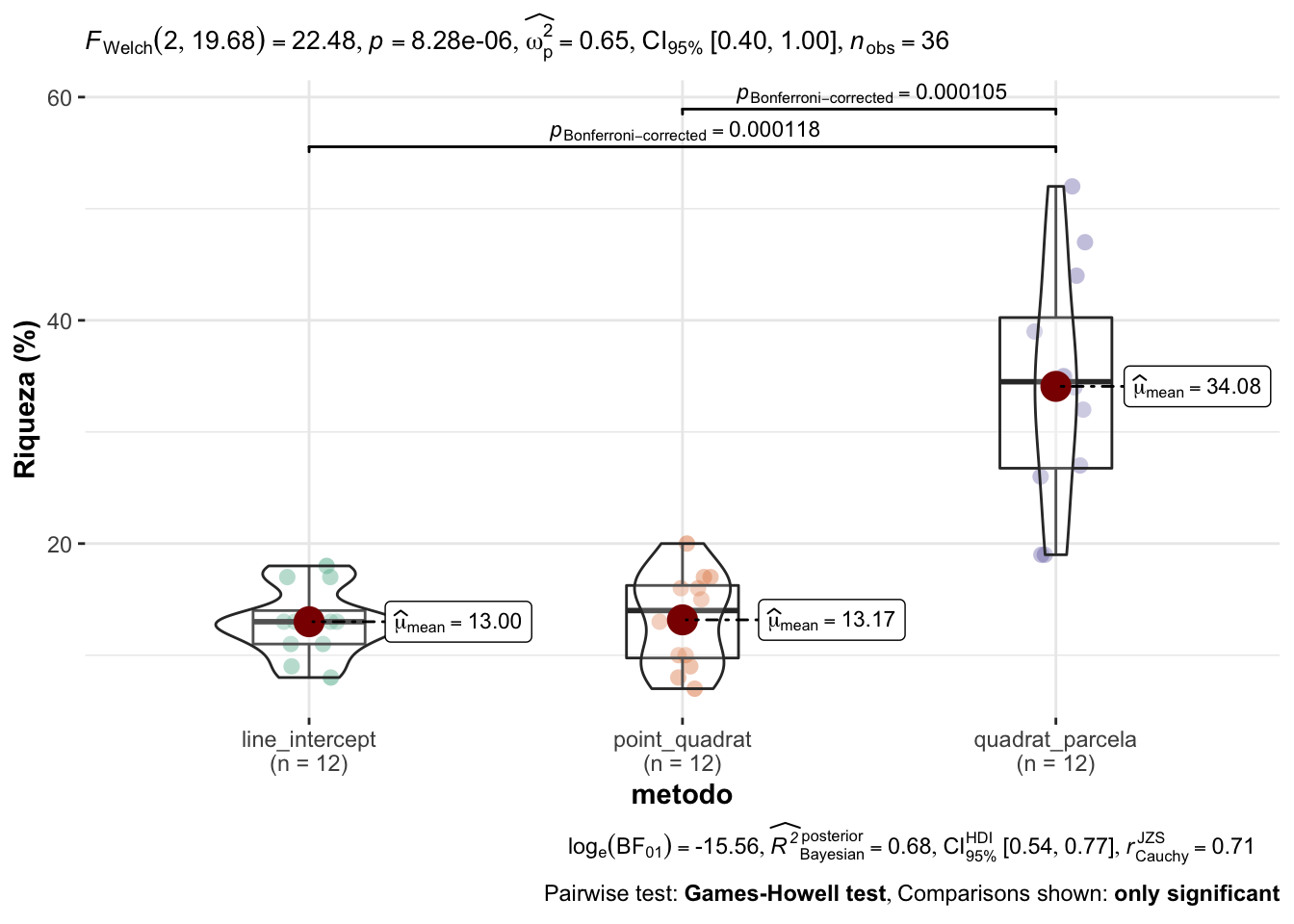
Figure 2.1: Comparación de los valores de riqueza entre los diferentes métodos de campo.
| Version | Author | Date |
|---|---|---|
| 3648c33 | ajpelu | 2022-04-19 |
2.1.3 Correlación
Tal y como observamos en la Figura 2.2, los métodos de line intercept y point quadrat, subestiman la riqueza estimada por el método quadrat parcela. Asimismo observamos una buena correlación entre la riqueza estimada por line intercept y point quadrat (Figura 2.3) (\(R^2=\) 0.66).
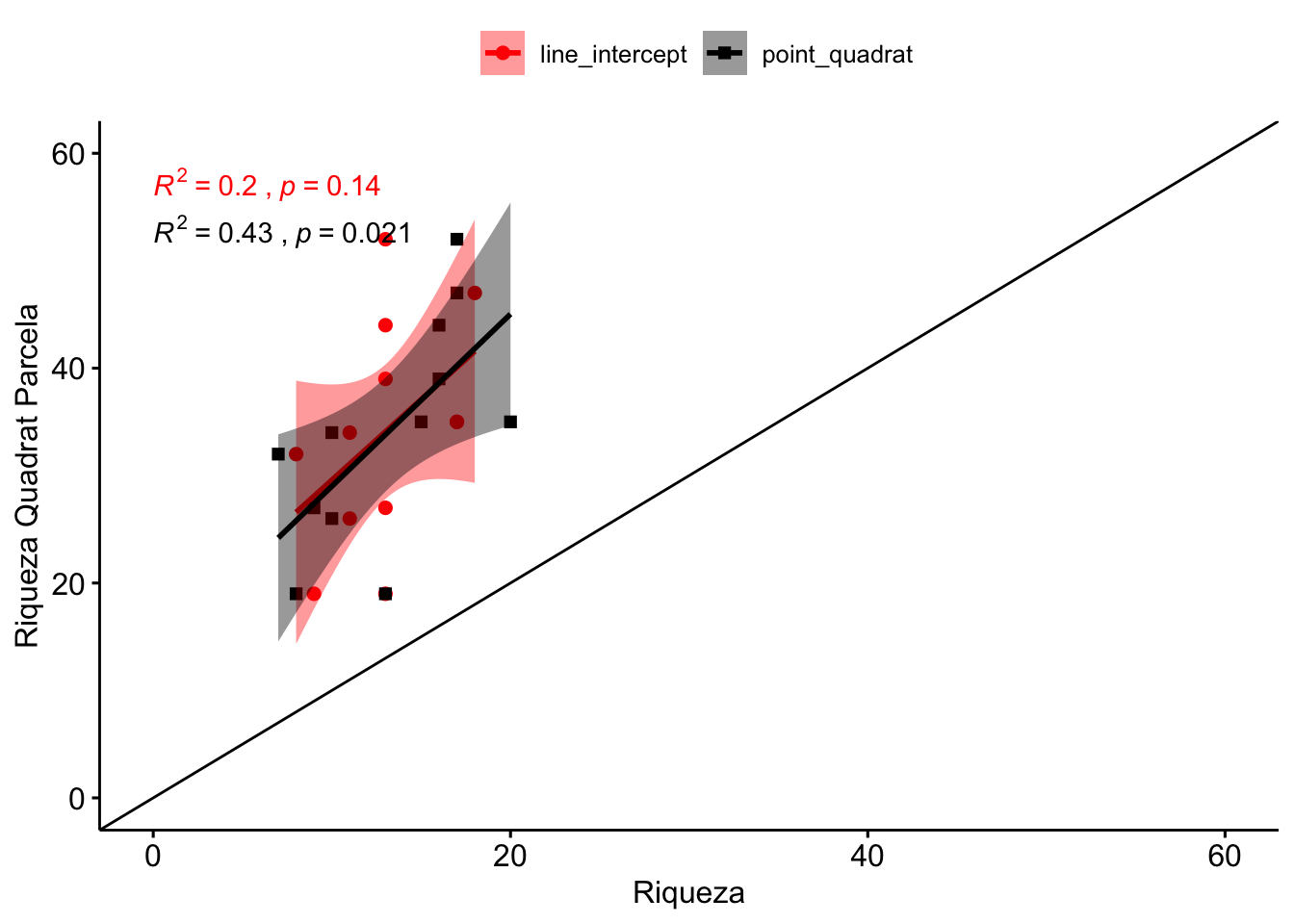
Figure 2.2: Correlación entre los valores de riqueza estimados por Quadrat Parcela y los otros métodos de campo: Line Intercept y Point Quadrat
2.1.3.1 Correlación Line Intercept - Point Quadrat
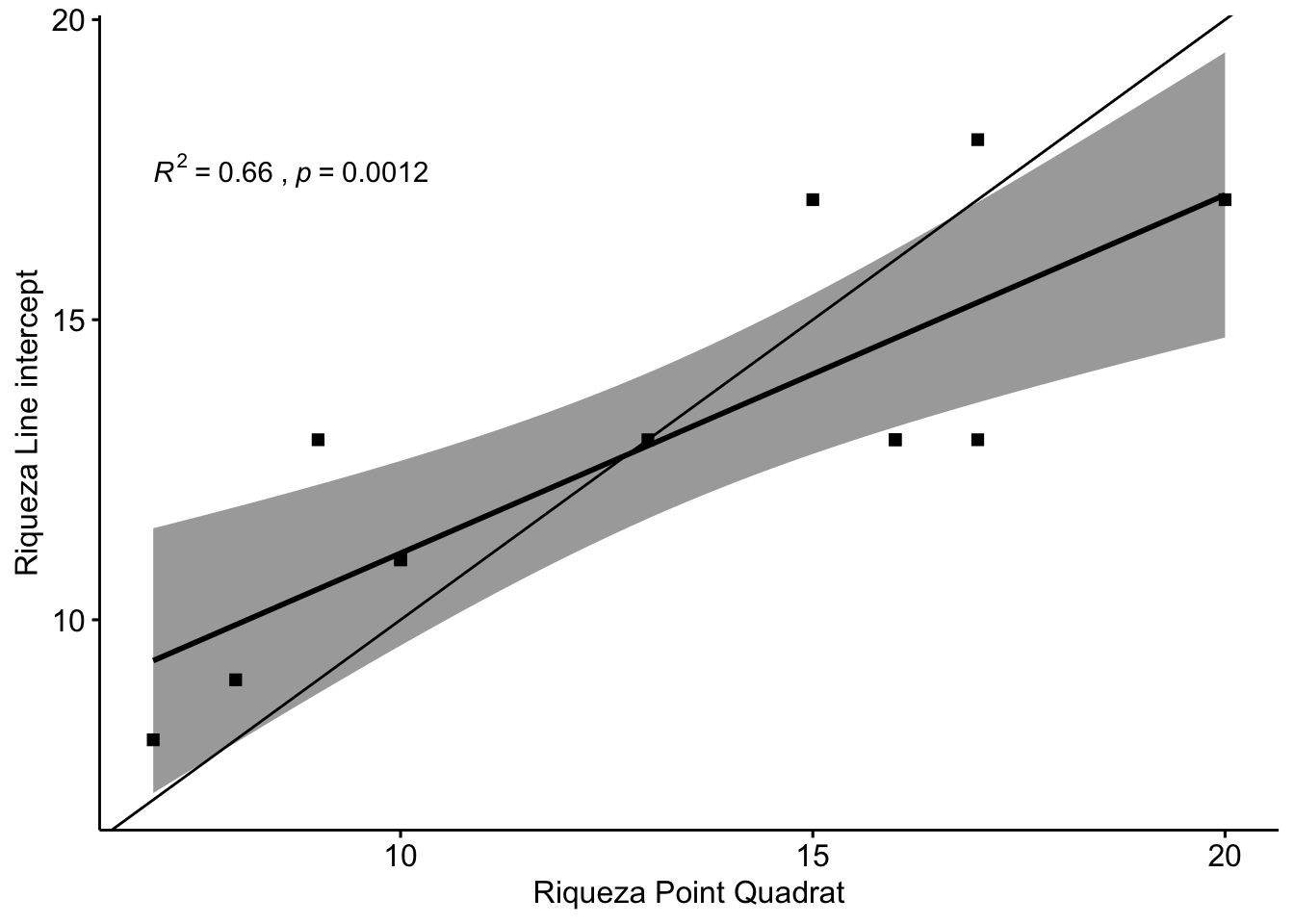
Figure 2.3: Correlación entre los valores de riqueza estimados por Line Intercept y Point Quadrat
2.2 Diversidad
Comparamos entre line intercept, quadrat medio y point quadrat.
2.2.1 Summary values
| metodo | mean | sd | se | cv | median | min | max | n |
|---|---|---|---|---|---|---|---|---|
| line_intercept | 1.72 | 0.49 | 0.14 | 28.71 | 1.63 | 1.10 | 2.80 | 12 |
| point_quadrat | 1.99 | 0.45 | 0.13 | 22.82 | 1.95 | 1.31 | 2.68 | 12 |
| quadrat medio | 1.34 | 0.20 | 0.06 | 14.78 | 1.38 | 0.97 | 1.58 | 12 |
2.2.2 Comparación de métodos
- ANOVA Kruskal Wallis
| statistic | p.value | parameter | method | mi_variable |
|---|---|---|---|---|
| 11.47748 | 0.0032188 | 2 | Kruskal-Wallis rank sum test | diversidad |
Los resultados de la ANOVA no paramétrica (Kruskal-Wallis) indican que existen diferencias significativas para la riqueza de especies vegetales entre los diferentes métodos de campo empleados (\(\chi^2\) = 11.48; p=0.0032) (Tabla 2.5).
Posteriormente, evaluamos si existen diferencias entre cada uno de los métodos (post hoc) y observamos que solamente existen diferencias significativas entre el point quadrat y el quadrat medio (Tabla 2.6, Figura 2.4).
| H0 | statistic | p.value |
|---|---|---|
| line_intercept = point_quadrat | 1.39 | 0.489 |
| line_intercept = quadrat medio | 1.98 | 0.144 |
| point_quadrat = quadrat medio | 3.37 | 0.002 |
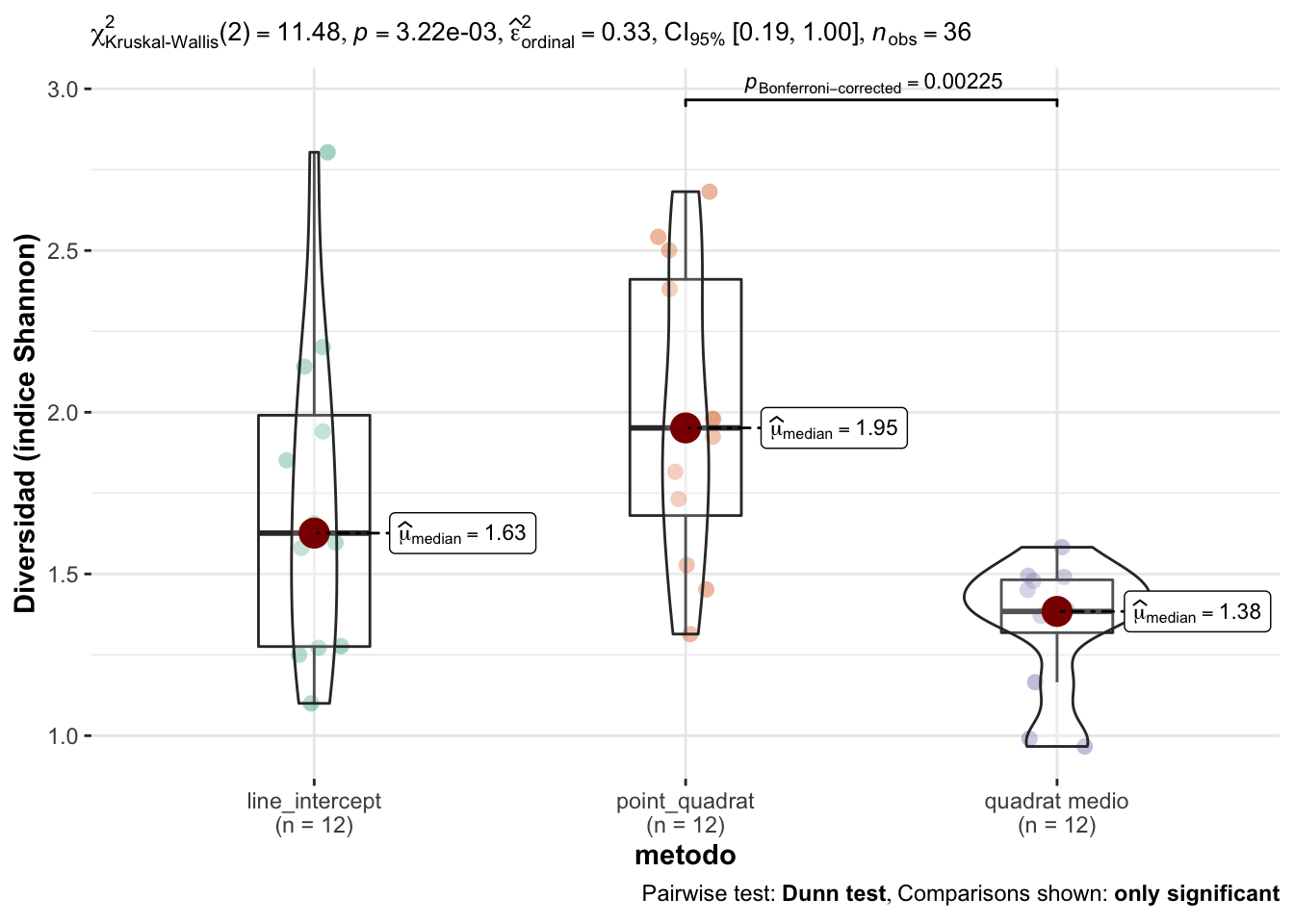
Figure 2.4: Comparación de los valores de diversidad (Shannon) entre los diferentes métodos de campo.
| Version | Author | Date |
|---|---|---|
| 3648c33 | ajpelu | 2022-04-19 |
2.2.3 Correlación
El método line intercept subestima la diversidad en comparación con el método point quadrat (Figura 2.5), aunque presentan una significativa y alta correlación (\(R^2=\) 0.82); mientras que el LI sobreestima la diversidad en comparación con el método quadrat medio. La correlación entre point quadrat y quadrat medio es baja (Figura 2.6).
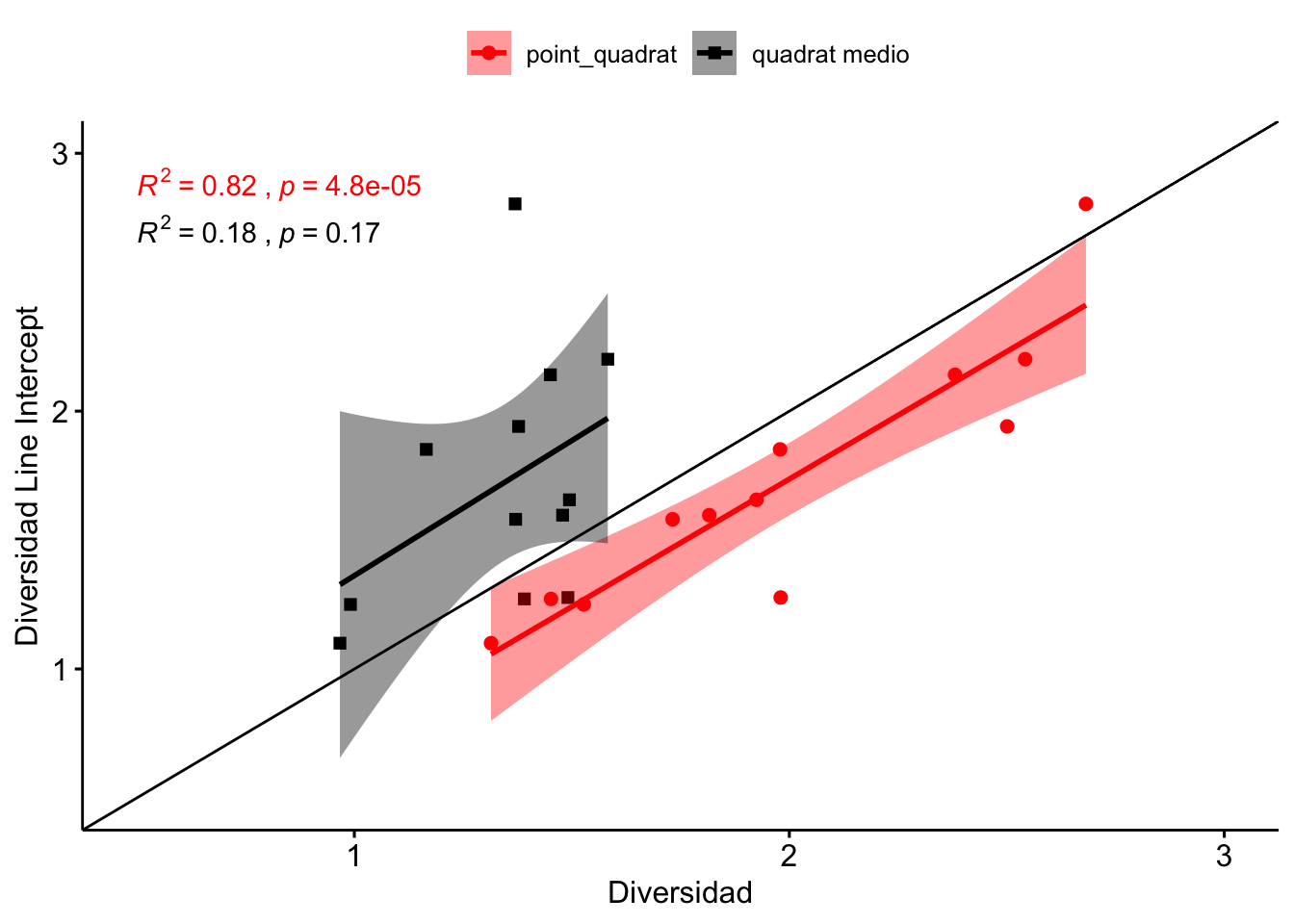
Figure 2.5: Correlación entre los valores de diversidad estimados por Line Intercept y los otros métodos de campo: Point Quadrat y Quadrat Medio
2.2.3.1 Correlación Quadrat Medio - Point Quadrat
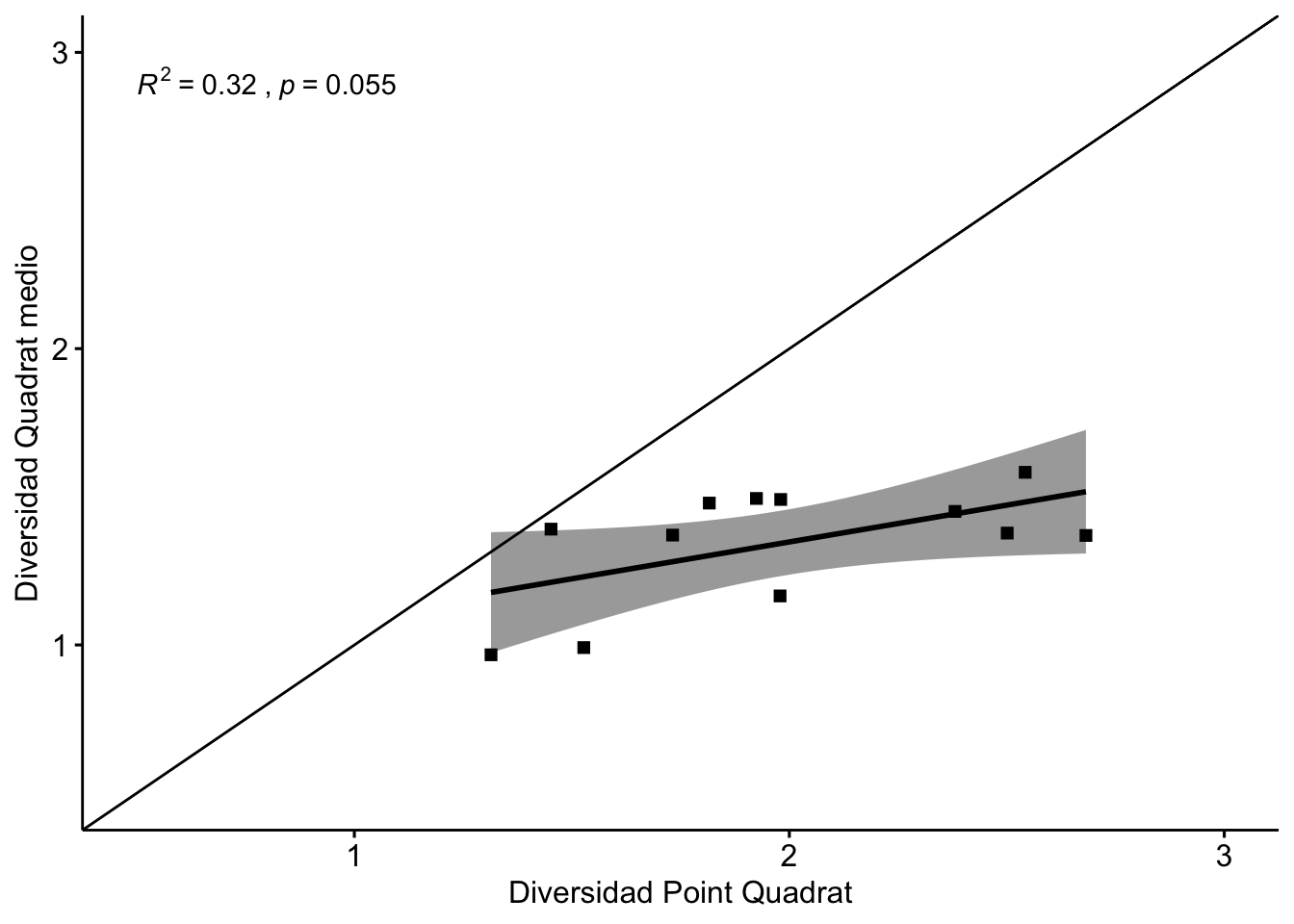
Figure 2.6: Correlación entre los valores de diversidad estimados por Quadrat Medio y Point Quadrat.
3 Objetivo 3. Tecnológico
3.1 Cobertura dronQ ~ quadrats
3.1.1 Summary values
| metodo | mean | sd | se | cv | median | min | max | n |
|---|---|---|---|---|---|---|---|---|
| dronQ | 23.55 | 21.01 | 2.14 | 89.23 | 15.74 | 0.96 | 92.56 | 96 |
| quadrat | 29.92 | 18.47 | 1.89 | 61.75 | 26.50 | 6.00 | 87.00 | 96 |
3.1.2 Comparación de métodos
Los datos no son normales (W = 0.89; p<0.0001); pero si se cumple la homocedasticidad (Bartlett’s K-squared = 1.56; p=0.2119). Aplicamos una comparación no paramétrica (Wilcoxon Test), y observamos que existen diferencias significativas entre los valores de cobertura estimados en los quadrats frente a los estimados por el dronQ (Tabla 3.2). La cobertura estimada por quadrat presentan valores de la mediana superiores a los estimados con dronQ (Figura 3.1).
| statistic | p.value | method | alternative |
|---|---|---|---|
| 3248.5 | <0.001 | Wilcoxon rank sum test with continuity correction | two.sided |
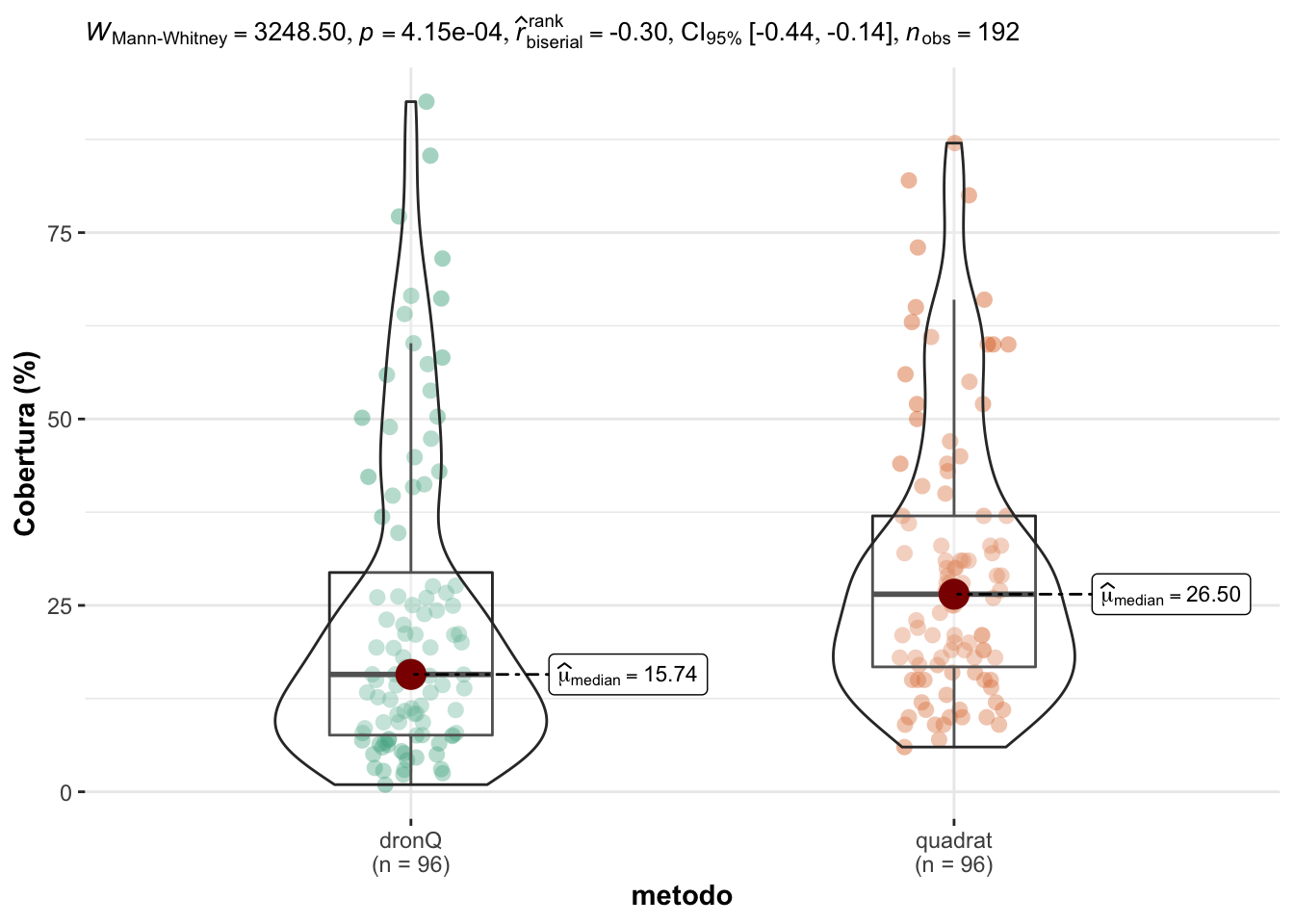
Figure 3.1: Comparación de los valores de cobertura entre DronQ y Quadrat.
3.1.3 Correlación
Observamos una alta correlación significativa entre los valores de cobertura estimados por dronQ y los estimados en campo por los quadrats (\(R^2=\) 0.82) (3.2, aunque observamos que el quadrat sobreestima los valores dados por el dronQ. Asimismo observamos un valor de RMSE de 10.86 (y un valor de RMSE normalizado de 13.41) (Tabla 3.3).
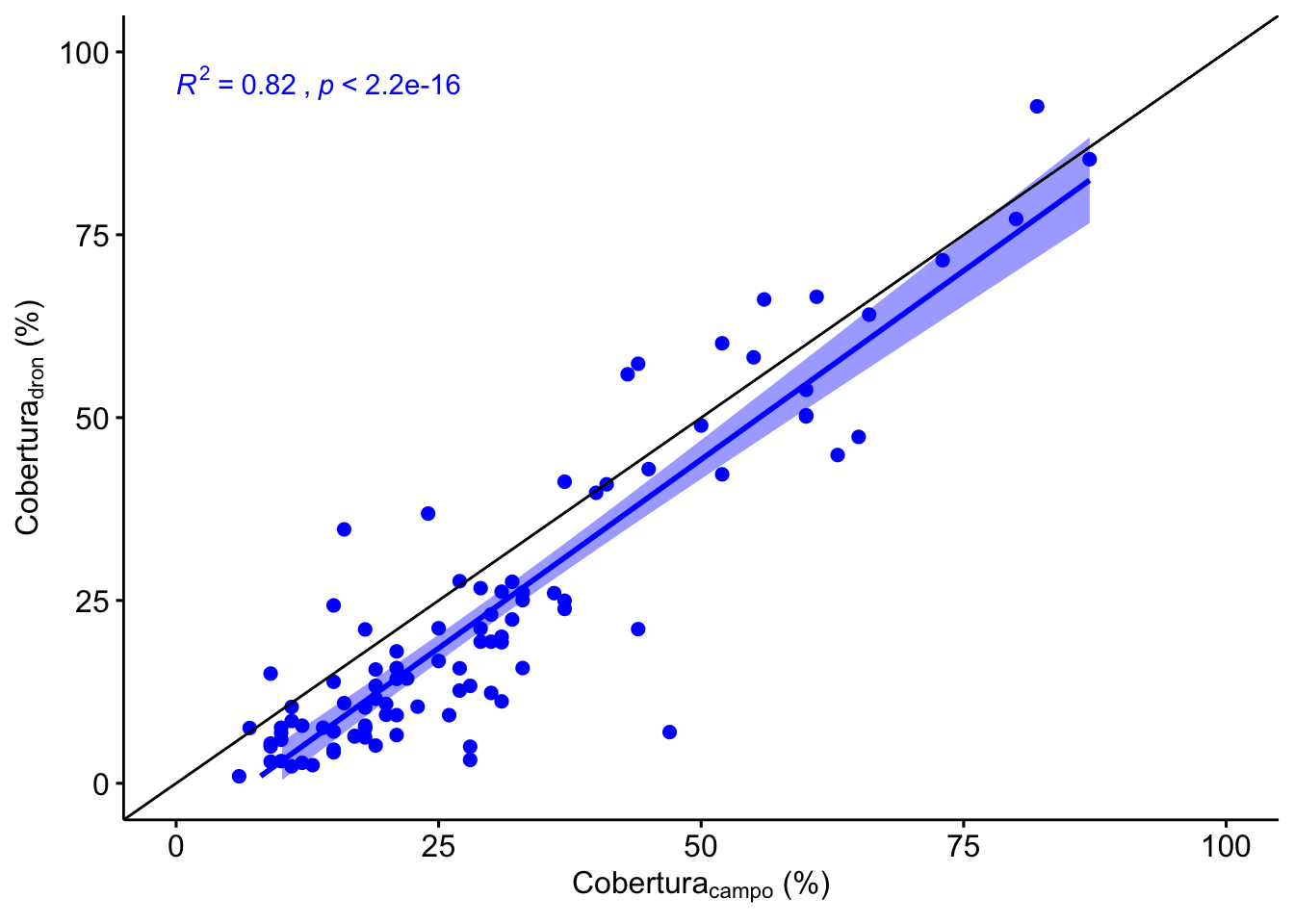
Figure 3.2: Correlación entre los valores de cobertura estimados por dronQ y Quadrat.
| rmse | min | max | rmsen.minmax |
|---|---|---|---|
| 10.86 | 6 | 87 | 13.41 |
3.1.3.1 Partición de la varianza
¿Es la relación entre dronQ-Quadrat uniforme a lo largo de todos los valores de cobertura o existen grupos dentro de esta correlación? Dicho de otro modo, ¿la correlación entre las dos variables es homogénea a lo largo de todos los valores de cobertura?. Para ello aplicamos una técnica de partición de la varianza, y hacemos árboles de clasificación.
Model formula:
dronQ ~ quadrat
Fitted party:
[1] root
| [2] quadrat <= 37
| | [3] quadrat <= 23
| | | [4] quadrat <= 14: 6.028 (n = 17, err = 199.7)
| | | [5] quadrat > 14: 11.864 (n = 27, err = 1197.2)
| | [6] quadrat > 23: 20.458 (n = 29, err = 1988.0)
| [7] quadrat > 37
| | [8] quadrat <= 52: 41.624 (n = 10, err = 2476.0)
| | [9] quadrat > 52: 63.698 (n = 13, err = 2668.2)
Number of inner nodes: 4
Number of terminal nodes: 5$`1`
quadrat
statistic 7.825365e+01
p.value 9.062347e-19
$`2`
quadrat
statistic 3.184656e+01
p.value 1.668448e-08
$`3`
quadrat
statistic 6.645925944
p.value 0.009938281
$`4`
NULL
$`5`
quadrat
statistic 0.01297757
p.value 0.90930188
$`6`
quadrat
statistic 3.81413871
p.value 0.05082173
$`7`
quadrat
statistic 1.231918e+01
p.value 4.483287e-04
$`8`
NULL
$`9`
NULLLos resultados indican Nos indica que existen al menos 5 grupos (Figura 3.3. Sin embargo, podemos estar sobreparametrizando. Para ello evaluamos si existe overfitting:
Hacer crecer un árbol sobre cierto nivel de complejidad, puede llevarnos a sobreparametrizar el modelo. ¿En que nodo cortamos el árbol?
Es preferible tener un modelo más simple que sobreparametrizar el ajuste. Para ello podemos utilizar el Complexity parameter (CP), que controla el tamaño del árbol de decisión. Si el coste de añadir otro nodo (u otra variable) a partir del nodo actual está por encima del valor del cp, entonces el árbol no continúa creciendo. Mas información aquí.
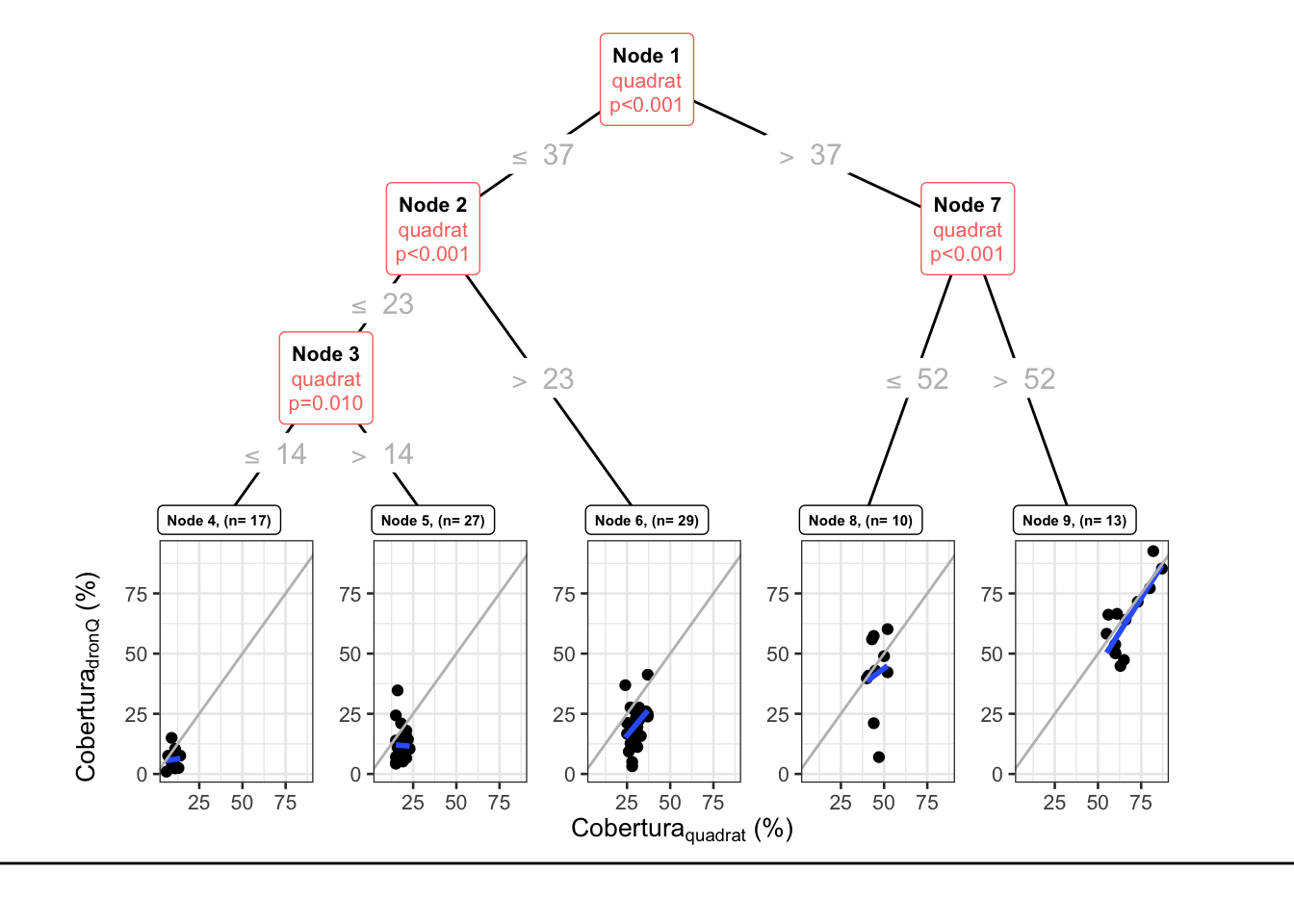
Figure 3.3: Relación entre la cobertura estimada con DronQ y con Quadrat. Partición de la varianza (modelo global)
Regression tree:
rpart(formula = dronQ ~ quadrat, data = dfcob_dronq)
Variables actually used in tree construction:
[1] quadrat
Root node error: 41934/96 = 436.82
n= 96
CP nsplit rel error xerror xstd
1 0.673397 0 1.00000 1.02574 0.179216
2 0.065679 1 0.32660 0.41939 0.092489
3 0.049060 2 0.26092 0.43811 0.088174
4 0.010000 3 0.21186 0.32548 0.065304| CP | nsplit | rel error | xerror | xstd |
|---|---|---|---|---|
| 0.6734 | 0 | 1.000 | 1.026 | 0.1792 |
| 0.0657 | 1 | 0.327 | 0.419 | 0.0925 |
| 0.0491 | 2 | 0.261 | 0.438 | 0.0882 |
| 0.0100 | 3 | 0.212 | 0.325 | 0.0653 |
Al explorar este modelo observamos que el error (xerror) (Tabla 3.4) no se disminuye sustancialmente a partir del 3 item (nsplit = 2) (Figura 3.4). Por tanto volvemos a modelar, usando un cp = 0.22.
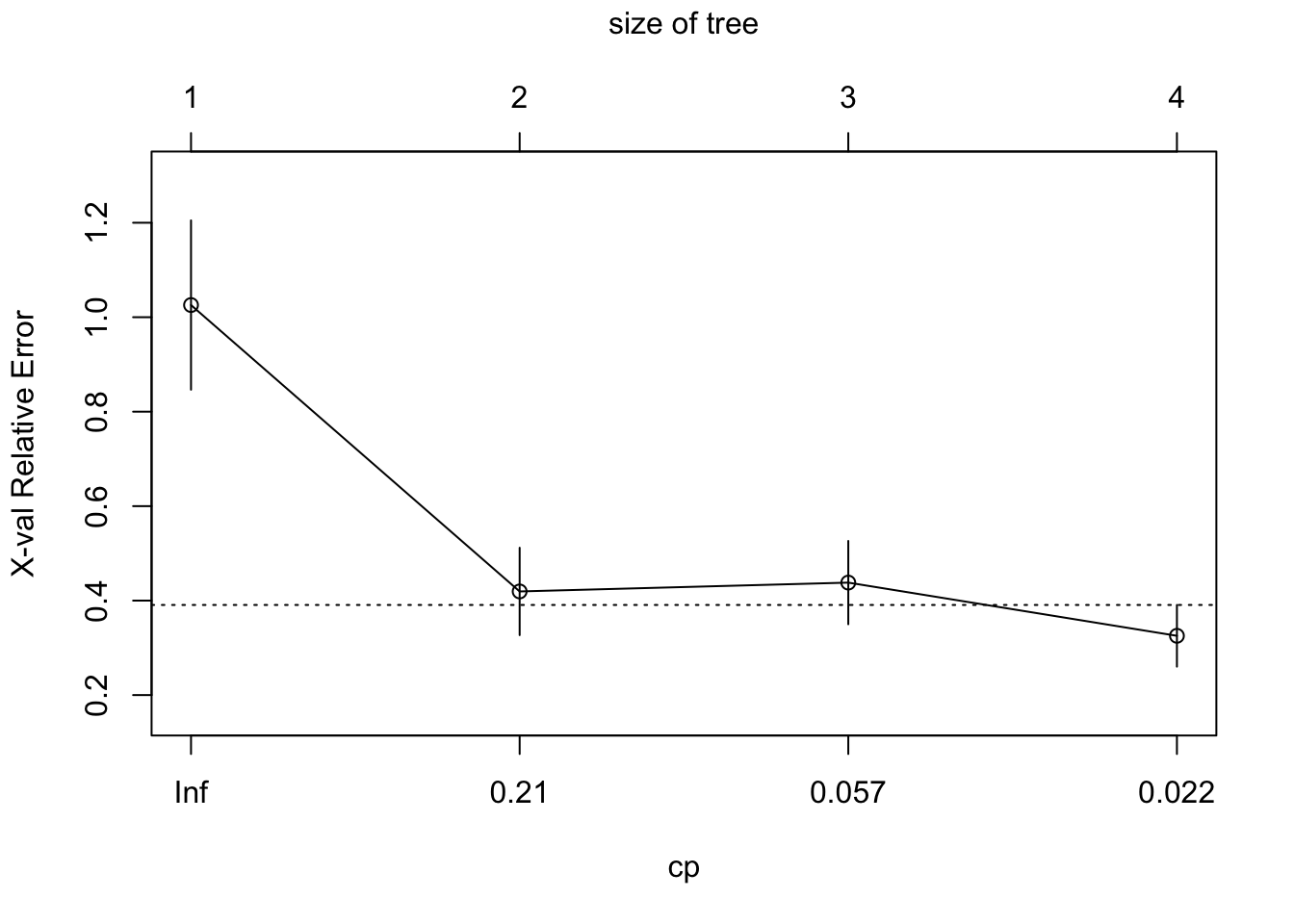
Figure 3.4: Error relativo frente a CP del modelo de partición de la varianza completo de la cobertura
Regression tree:
rpart(formula = dronQ ~ quadrat, data = dfcob_dronq, control = rpart.control(minsplit = 2,
cp = 0.22))
Variables actually used in tree construction:
[1] quadrat
Root node error: 41934/96 = 436.82
n= 96
CP nsplit rel error xerror xstd
1 0.6734 0 1.0000 1.02574 0.179216
2 0.2200 1 0.3266 0.41939 0.092489| CP | nsplit | rel error | xerror | xstd |
|---|---|---|---|---|
| 0.6734 | 0 | 1.000 | 1.026 | 0.1792 |
| 0.2200 | 1 | 0.327 | 0.419 | 0.0925 |
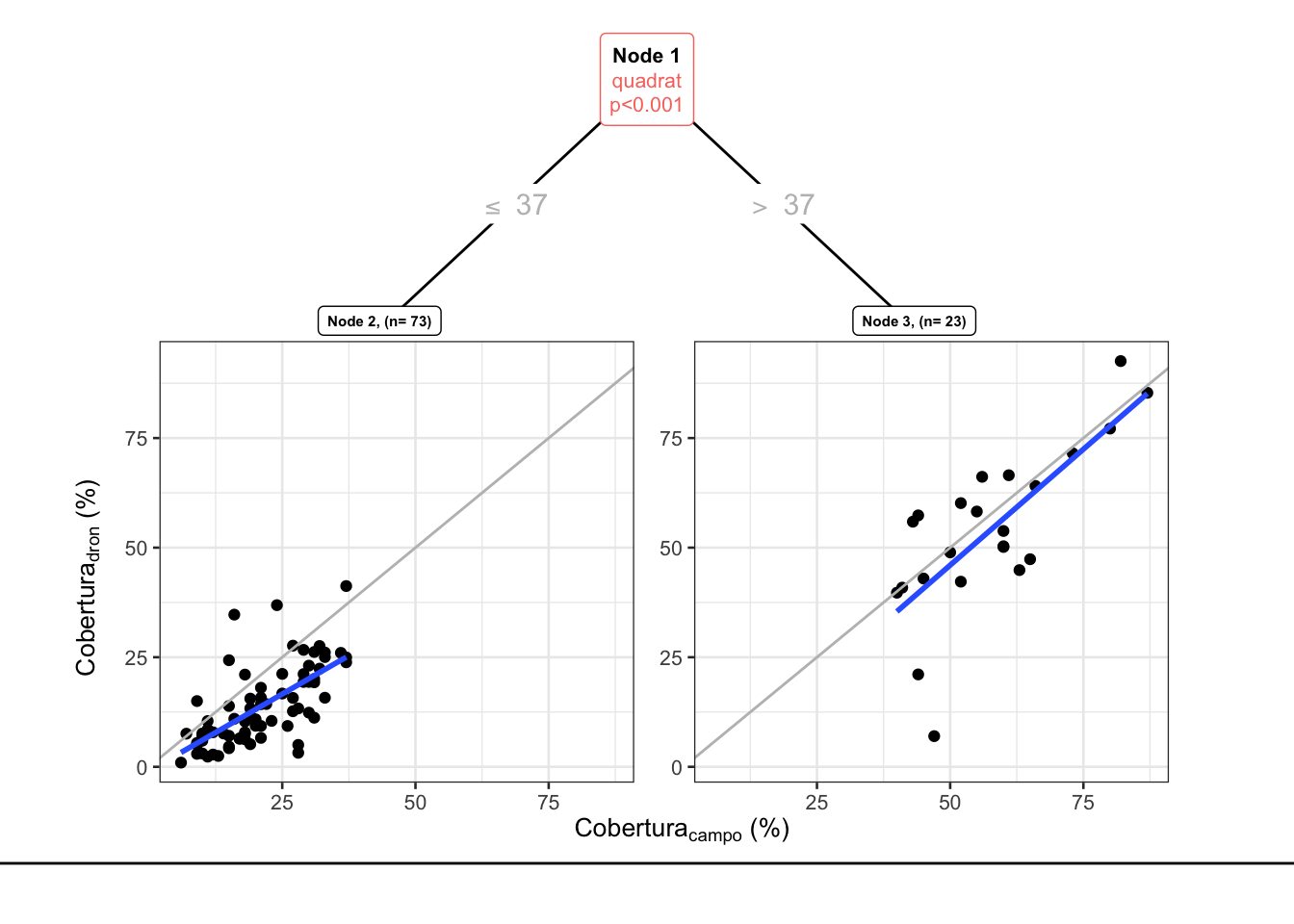
Figure 3.5: Relación entre la cobertura estimada con DronQ y con Quadrat. Partición de la varianza (modelo final)
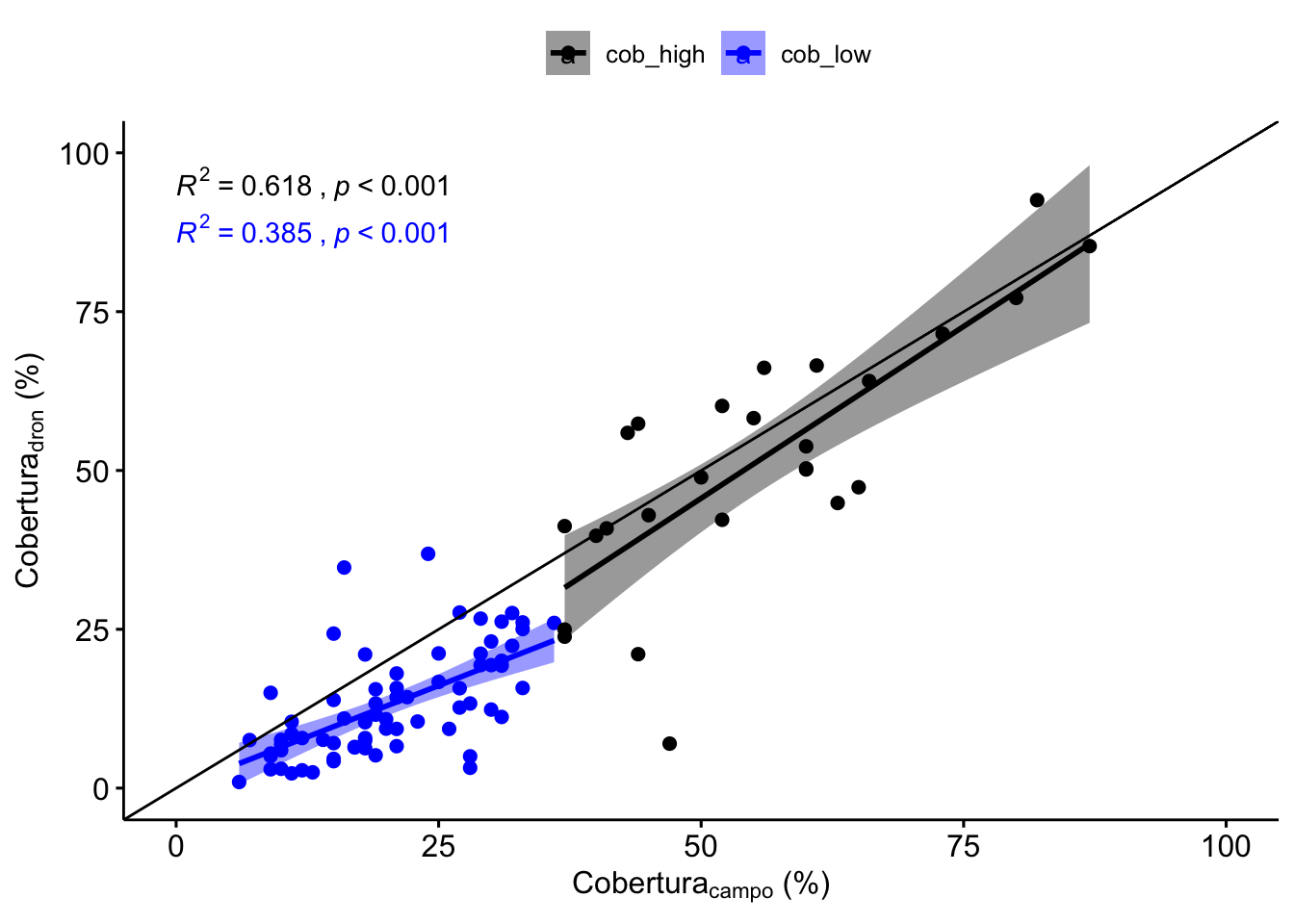
Figure 3.6: Correlación entre los valores de cobertura estimados por dronQ y Quadrat, considerando dos grupos (Coberturas inferiores (azul) y superiores (negro) a 36 % (en campo).
| clase | rmse | min | max | rmsen.minmax |
|---|---|---|---|---|
| cob_high | 12.58 | 37 | 87 | 25.16 |
| cob_low | 10.15 | 6 | 36 | 33.82 |
3.2 Cobertura dronT ~ LI y PQ
3.2.1 Summary values
| metodo | mean | sd | se | cv | median | min | max | n |
|---|---|---|---|---|---|---|---|---|
| line_intercept | 27.19 | 6.49 | 1.87 | 23.86 | 29.67 | 11.95 | 34.00 | 12 |
| point_quadrat | 55.50 | 7.62 | 2.20 | 13.73 | 56.00 | 41.00 | 64.00 | 12 |
| dronT | 17.46 | 2.71 | 0.78 | 15.51 | 17.07 | 14.29 | 22.92 | 12 |
3.2.2 Comparación de métodos
- ANOVA Kruskal Wallis
| statistic | p.value | parameter | method | mi_variable |
|---|---|---|---|---|
| 27.90057 | 9e-07 | 2 | Kruskal-Wallis rank sum test | cobertura |
Los resultados de la ANOVA no paramétrica (Kruskal-Wallis) indican que existen diferencias significativas para la cobertura (%) entre los diferentes métodos de campo empleados (\(\chi^2\) = 27.9; p<0.0001) (Tabla 3.8).
Posteriormente, evaluamos si existen diferencias entre cada uno de los métodos (post hoc) y observamos que existen diferencias significativas entre el point quadrat y los otros métodos (line intercept y dron Parcela) (Tabla 3.9, Figura 3.7). Asimismo, no observamos diferencias entre la cobertura estimada según el line intercept y el dron Parcela.
| H0 | statistic | p.value |
|---|---|---|
| line_intercept = point_quadrat | 3.12 | 0.005 |
| line_intercept = dronT | 2.13 | 0.099 |
| point_quadrat = dronT | 5.25 | <0.001 |
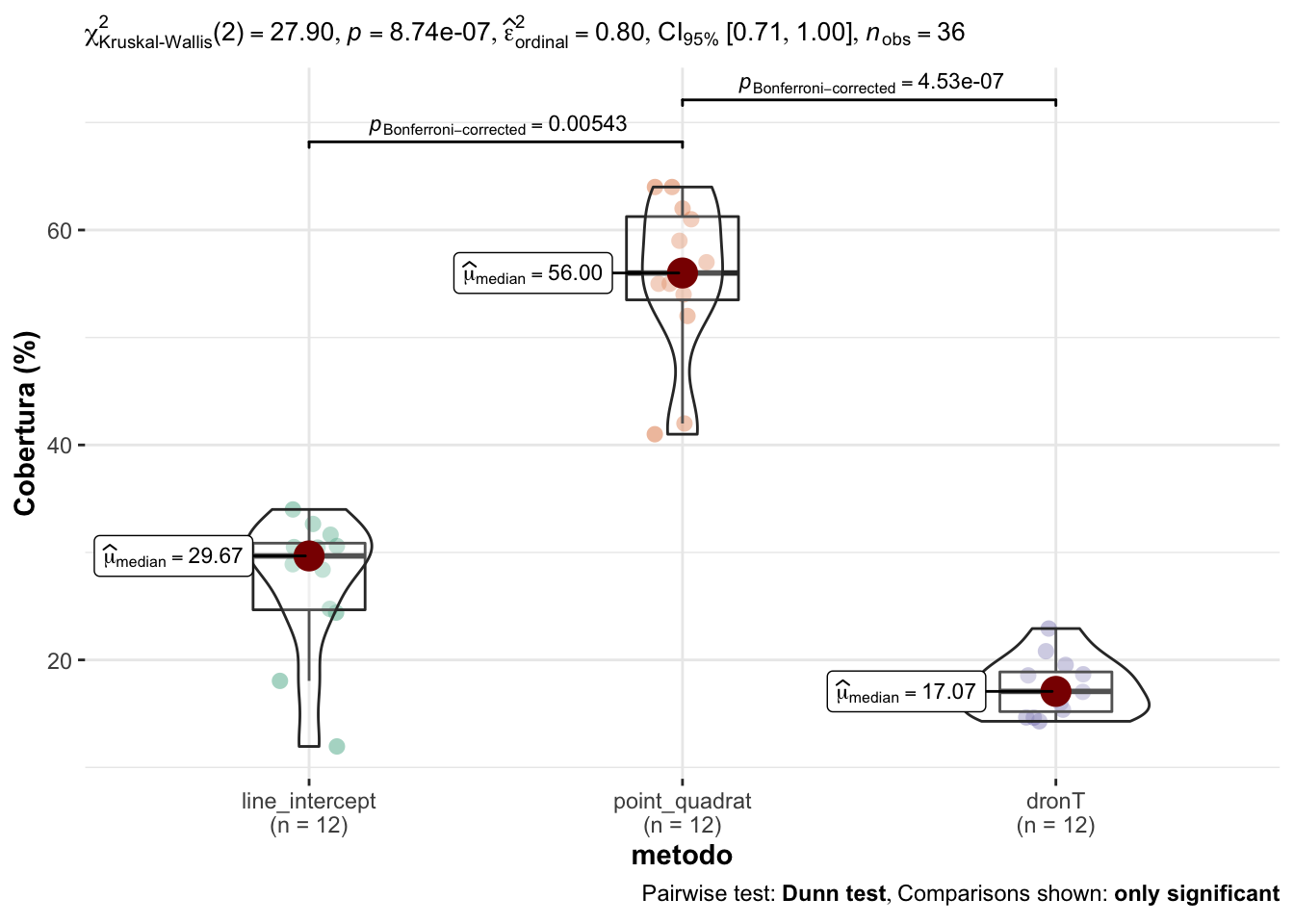
Figure 3.7: Comparación de los valores de cobertura entre los diferentes métodos de campo.
3.2.3 Correlación
Tanto line intercept como point quadrat subestiman los valores de cobertura dados por el dron en la parcela de 500 m^2 (Figura 3.8). Independientemente no presentan buenas correlaciones con el valor de cobertura estimado por el dron para toda la parcela.
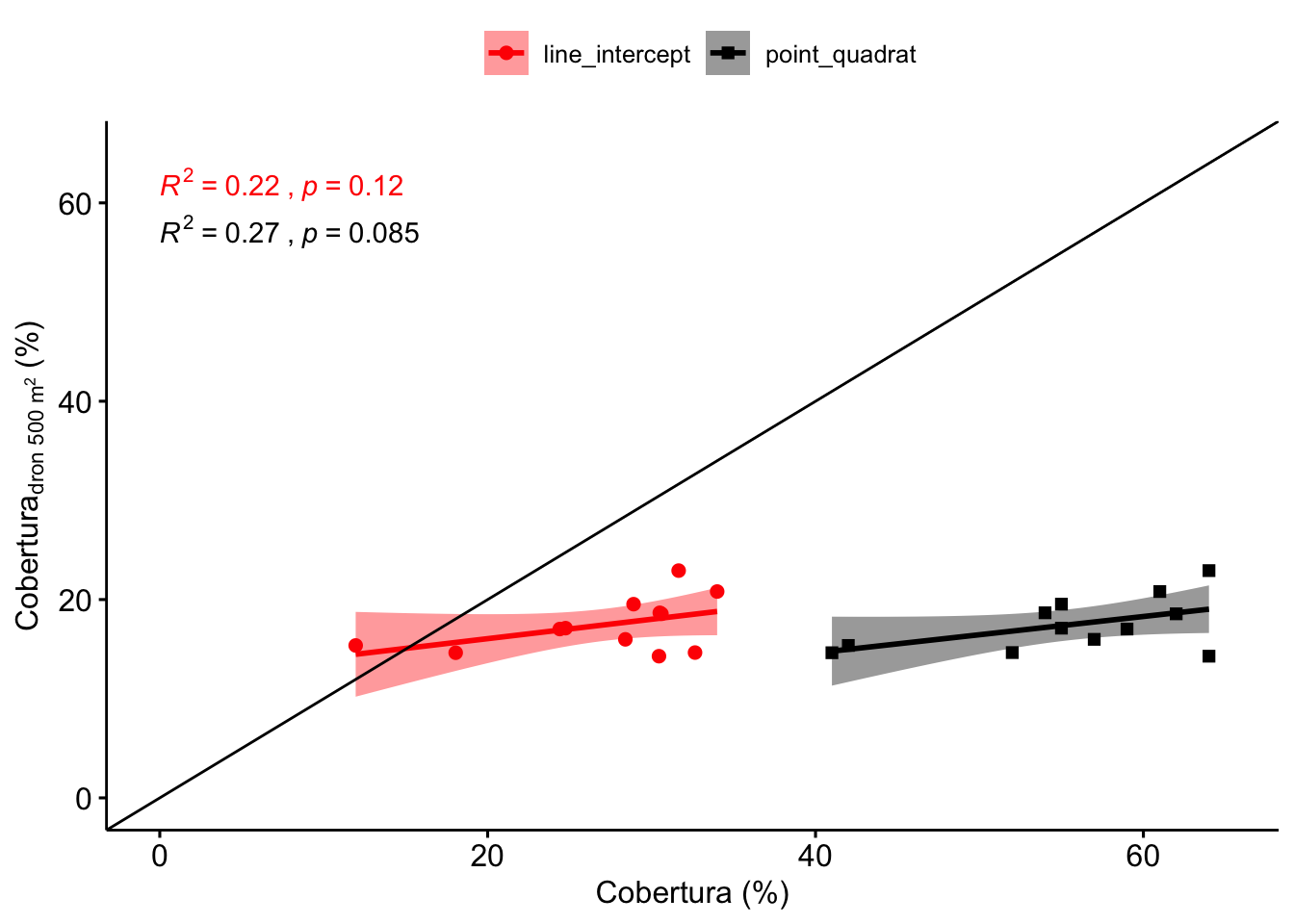
Figure 3.8: Correlación entre los valores de cobertura estimados por dronParcela con respecto a Line Intercept y Point Quadrat.
3.3 Fitovolumen dronQ ~ quadrats
3.3.1 Summary values
| metodo | mean | sd | se | cv | median | min | max | n |
|---|---|---|---|---|---|---|---|---|
| dronQ | 421.70 | 678.47 | 69.25 | 160.89 | 125.9 | 1.75 | 4077.36 | 96 |
| quadrat | 778.57 | 1108.78 | 113.16 | 142.41 | 323.4 | 28.05 | 5690.30 | 96 |
3.3.2 Comparación de métodos
Los datos no son normales (W = 0.63; p<0.0001) ni homocedasticos (Bartlett’s K-squared = 21.94; p<0.0001). Aplicamos una comparación no paramétrica (Wilcoxon Test), y observamos que existen diferencias significativas entre los valores de fitovolumen estimados en los quadrats frente a los estimados por el dronQ (Tabla 3.11). La cobertura estimada por quadrat presentan valores de la mediana superiores a los estimados con dronQ (Figura 3.9).
| statistic | p.value | method | alternative |
|---|---|---|---|
| 2965 | <0.001 | Wilcoxon rank sum test with continuity correction | two.sided |
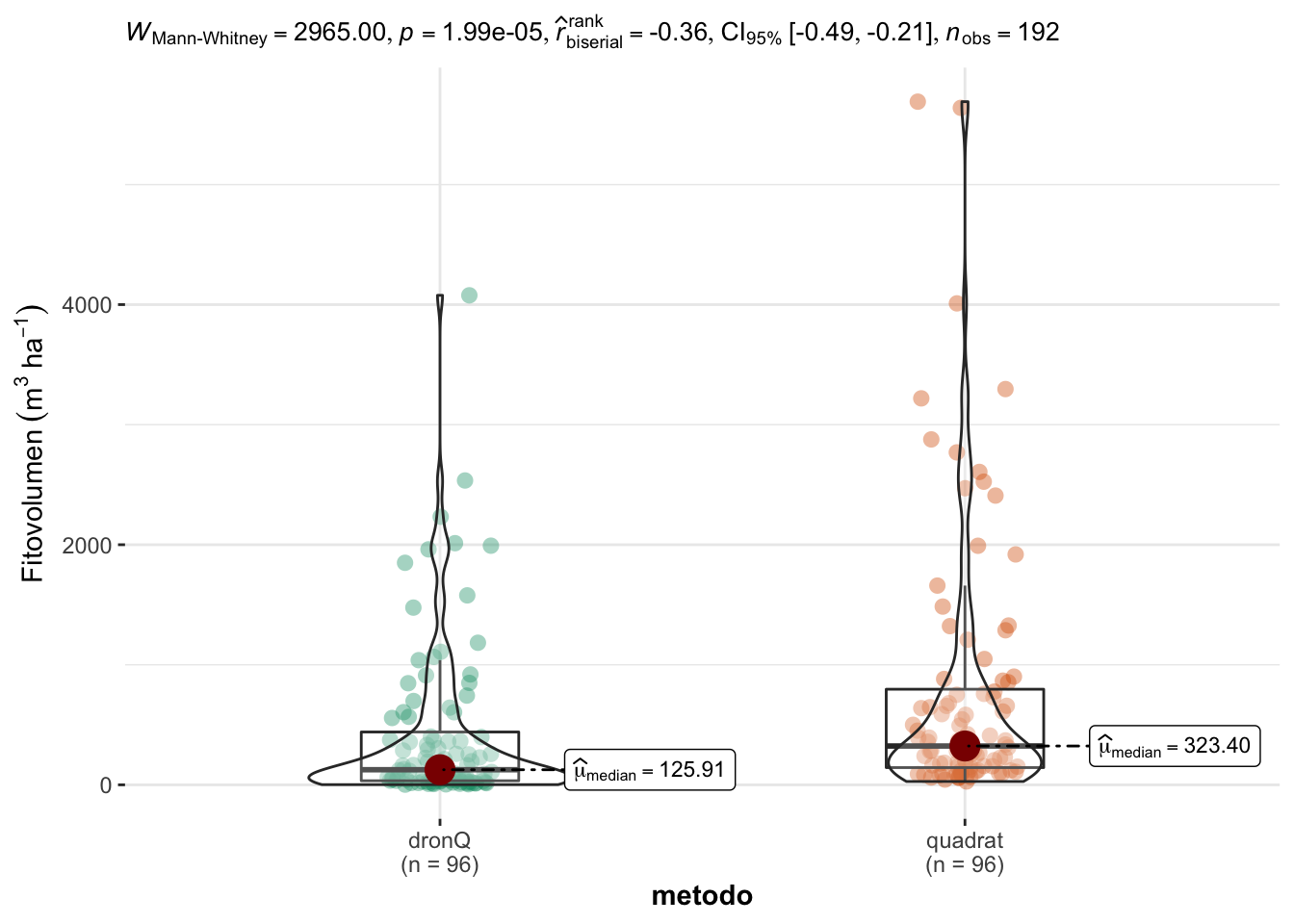
Figure 3.9: Comparación de los valores de fitovolumen entre DronQ y Quadrat.
3.3.3 Correlación
Observamos una alta correlación significativa entre los valores de fitovolumen estimados por dronQ y los estimados en campo por los quadrats (\(R^2=\) 0.9) (3.10, aunque observamos que el quadrat sobreestima los valores dados por el dronQ. Los valores de RMSE son de 619.49 (y un valor de RMSE normalizado de 10.94) (Tabla 3.12).
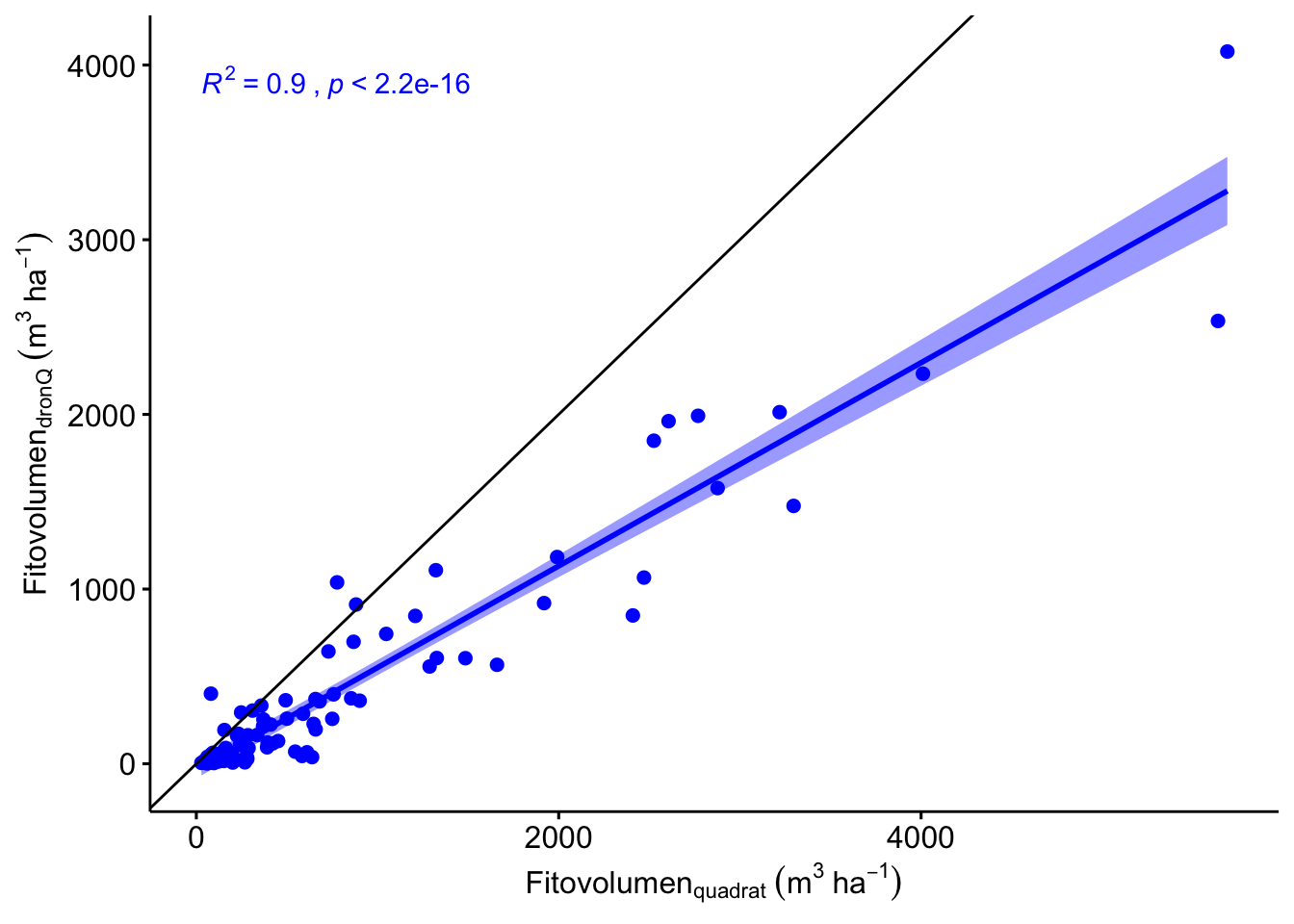
Figure 3.10: Correlación entre los valores de fitovolumen estimados por dronQ y Quadrat.
| rmse | min | max | rmsen.minmax |
|---|---|---|---|
| 619.49 | 28 | 5690 | 10.94 |
3.3.3.1 Partición de la varianza
¿Es la relación entre dronQ-Quadrat uniforme a lo largo de todos los valores de cobertura o existen grupos dentro de esta correlación? Dicho de otro modo, ¿la correlación entre las dos variables es homogénea a lo largo de todos los valores de cobertura?. Para ello aplicamos una técnica de partición de la varianza, y hacemos árboles de clasificación.
Model formula:
dronQ ~ quadrat
Fitted party:
[1] root
| [2] quadrat <= 2470.6
| | [3] quadrat <= 680.3
| | | [4] quadrat <= 288.3: 59.556 (n = 47, err = 276365.7)
| | | [5] quadrat > 288.3: 201.746 (n = 21, err = 240180.9)
| | [6] quadrat > 680.3: 722.725 (n = 19, err = 1345456.4)
| [7] quadrat > 2470.6: 2190.586 (n = 9, err = 4805690.0)
Number of inner nodes: 3
Number of terminal nodes: 4$`1`
quadrat
statistic 8.589206e+01
p.value 1.900300e-20
$`2`
quadrat
statistic 6.339813e+01
p.value 1.688783e-15
$`3`
quadrat
statistic 2.170388e+01
p.value 3.181471e-06
$`4`
quadrat
statistic 3.03838023
p.value 0.08131702
$`5`
quadrat
statistic 0.006282002
p.value 0.936826555
$`6`
NULL
$`7`
NULLLos resultados indican Nos indica que existen al menos 4 grupos (Figura 3.11. Sin embargo, podemos estar sobreparametrizando. Para ello evaluamos si existe overfitting:
Hacer crecer un árbol sobre cierto nivel de complejidad, puede llevarnos a sobreparametrizar el modelo. ¿En que nodo cortamos el árbol?
Es preferible tener un modelo más simple que sobreparametrizar el ajuste. Para ello podemos utilizar el Complexity parameter (CP), que controla el tamaño del árbol de decisión. Si el coste de añadir otro nodo (u otra variable) a partir del nodo actual está por encima del valor del cp, entonces el árbol no continúa creciendo. Mas información aquí.
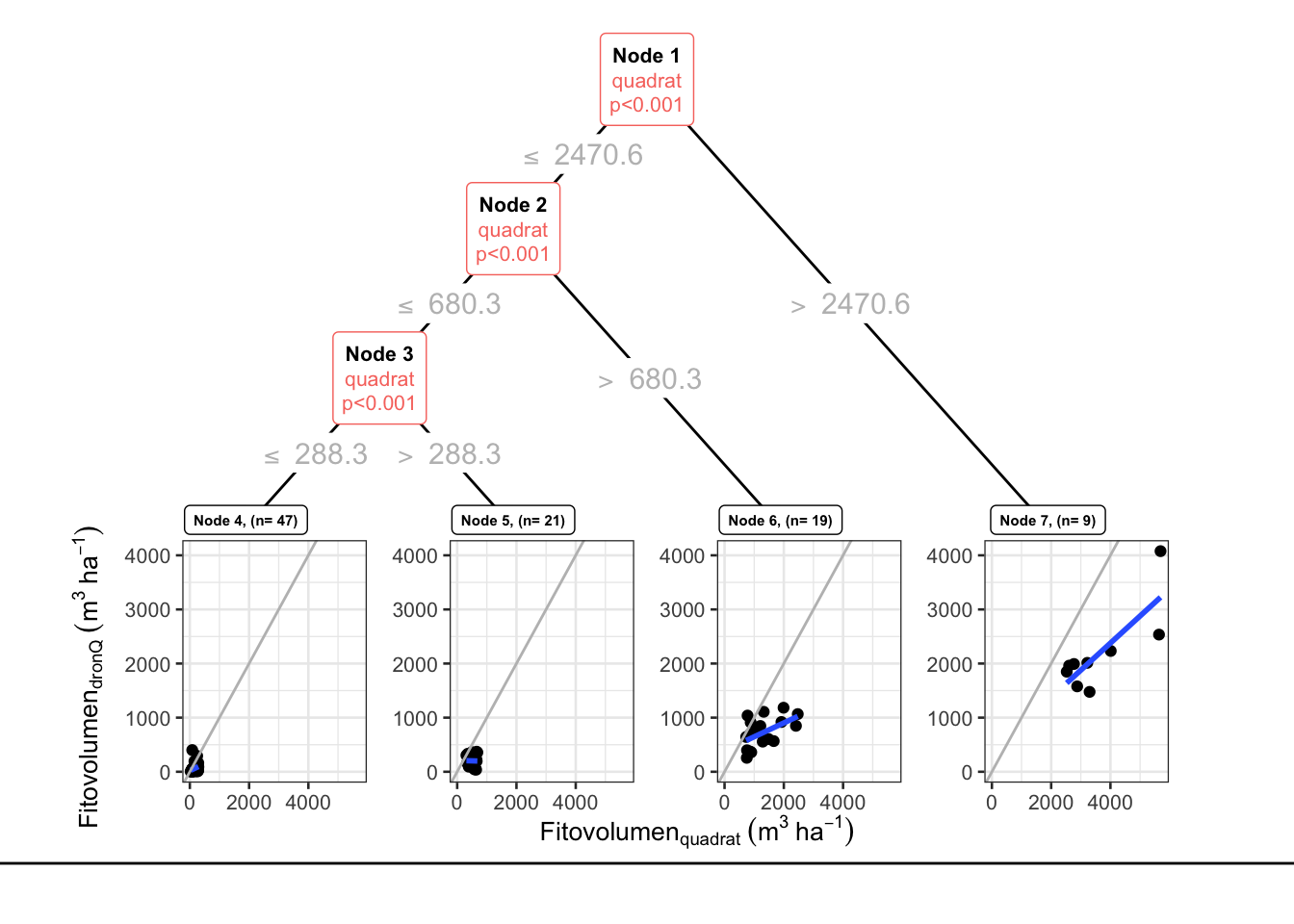
Figure 3.11: Relación entre el fitovolumen estimado con DronQ y con Quadrat. Partición de la varianza (modelo global)
Regression tree:
rpart(formula = dronQ ~ quadrat, data = dffit_dronq)
Variables actually used in tree construction:
[1] quadrat
Root node error: 43729935/96 = 455520
n= 96
CP nsplit rel error xerror xstd
1 0.71059 0 1.00000 1.01996 0.34309
2 0.13023 1 0.28941 0.42441 0.14781
3 0.01000 2 0.15919 0.30462 0.13893| CP | nsplit | rel error | xerror | xstd |
|---|---|---|---|---|
| 0.7106 | 0 | 1.000 | 1.020 | 0.3431 |
| 0.1302 | 1 | 0.289 | 0.424 | 0.1478 |
| 0.0100 | 2 | 0.159 | 0.305 | 0.1389 |
Al explorar este modelo observamos que el error (xerror) (Tabla 3.13) no se disminuye sustancialmente a partir del 2 item (nsplit = 1) (Figura 3.12). Por tanto volvemos a modelar, usando un cp = 0.3.
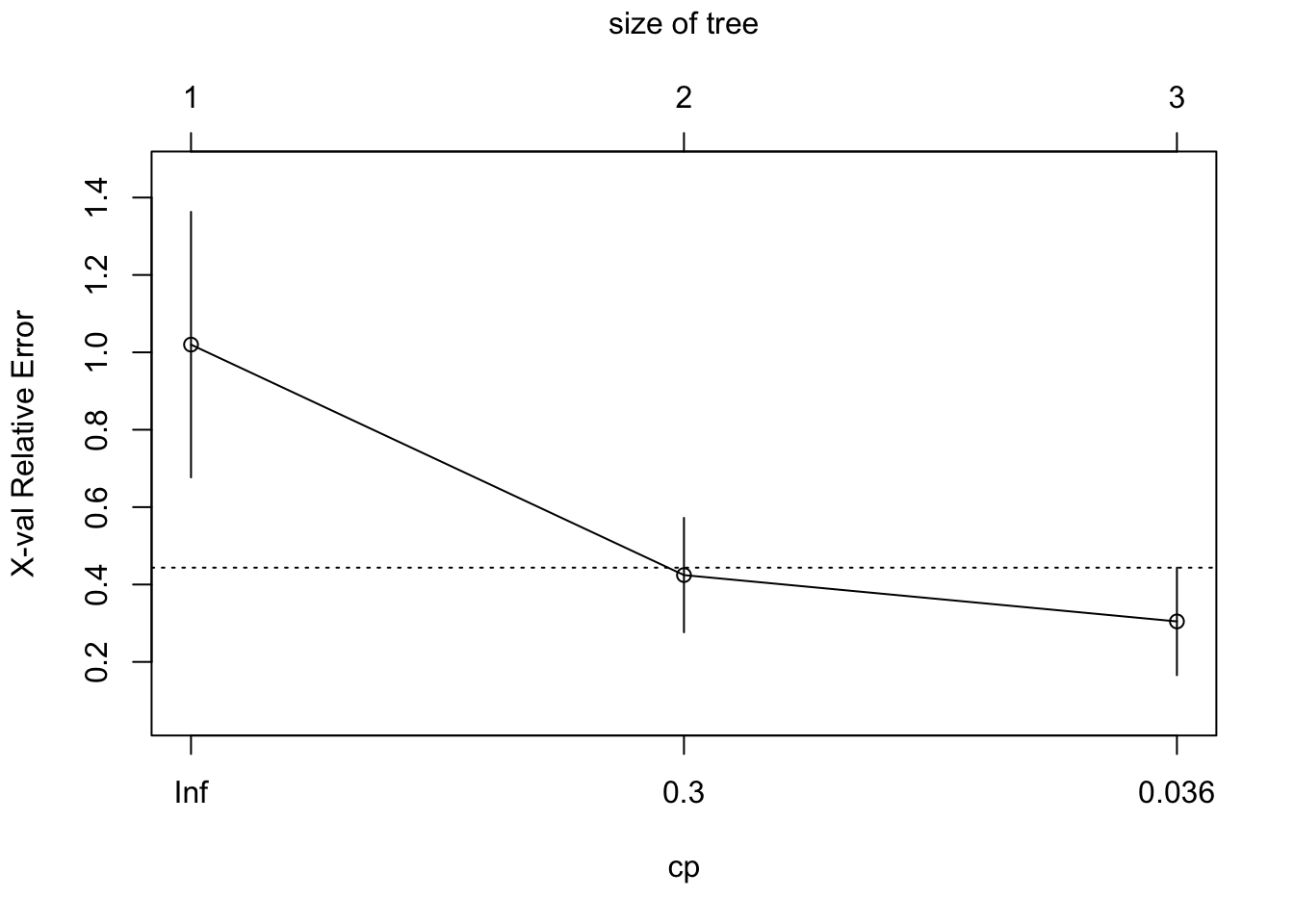
Figure 3.12: Error relativo frente a CP del modelo de partición de la varianza completo del fitovolumen
Regression tree:
rpart(formula = dronQ ~ quadrat, data = dffit_dronq, control = rpart.control(minsplit = 2,
cp = 0.3))
Variables actually used in tree construction:
[1] quadrat
Root node error: 43729935/96 = 455520
n= 96
CP nsplit rel error xerror xstd
1 0.71059 0 1.00000 1.01996 0.34309
2 0.30000 1 0.28941 0.42441 0.14781| CP | nsplit | rel error | xerror | xstd |
|---|---|---|---|---|
| 0.7106 | 0 | 1.000 | 1.020 | 0.3431 |
| 0.3000 | 1 | 0.289 | 0.424 | 0.1478 |
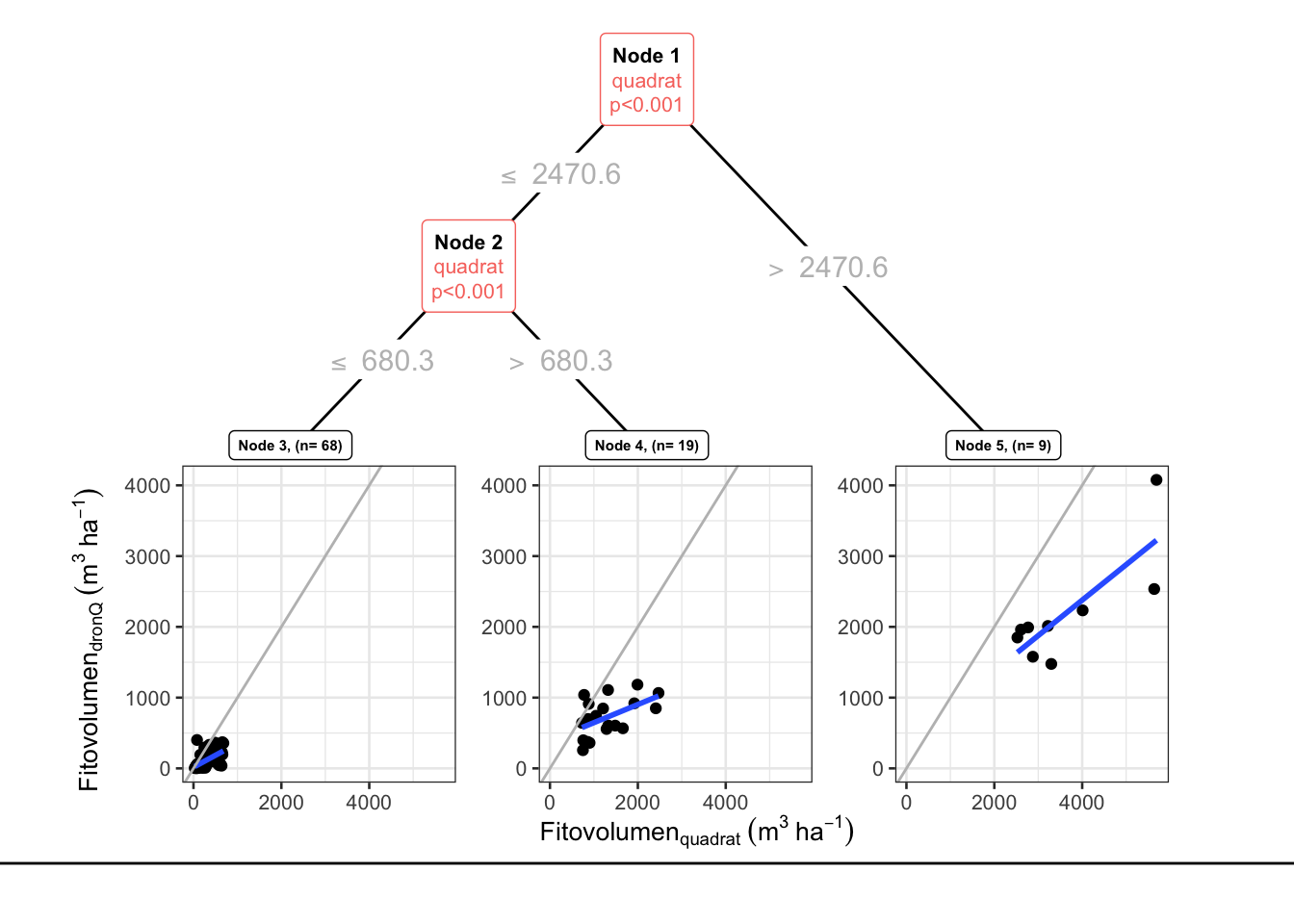
Figure 3.13: Relación entre el fitovolumen estimado con DronQ y con Quadrat. Partición de la varianza (modelo final)
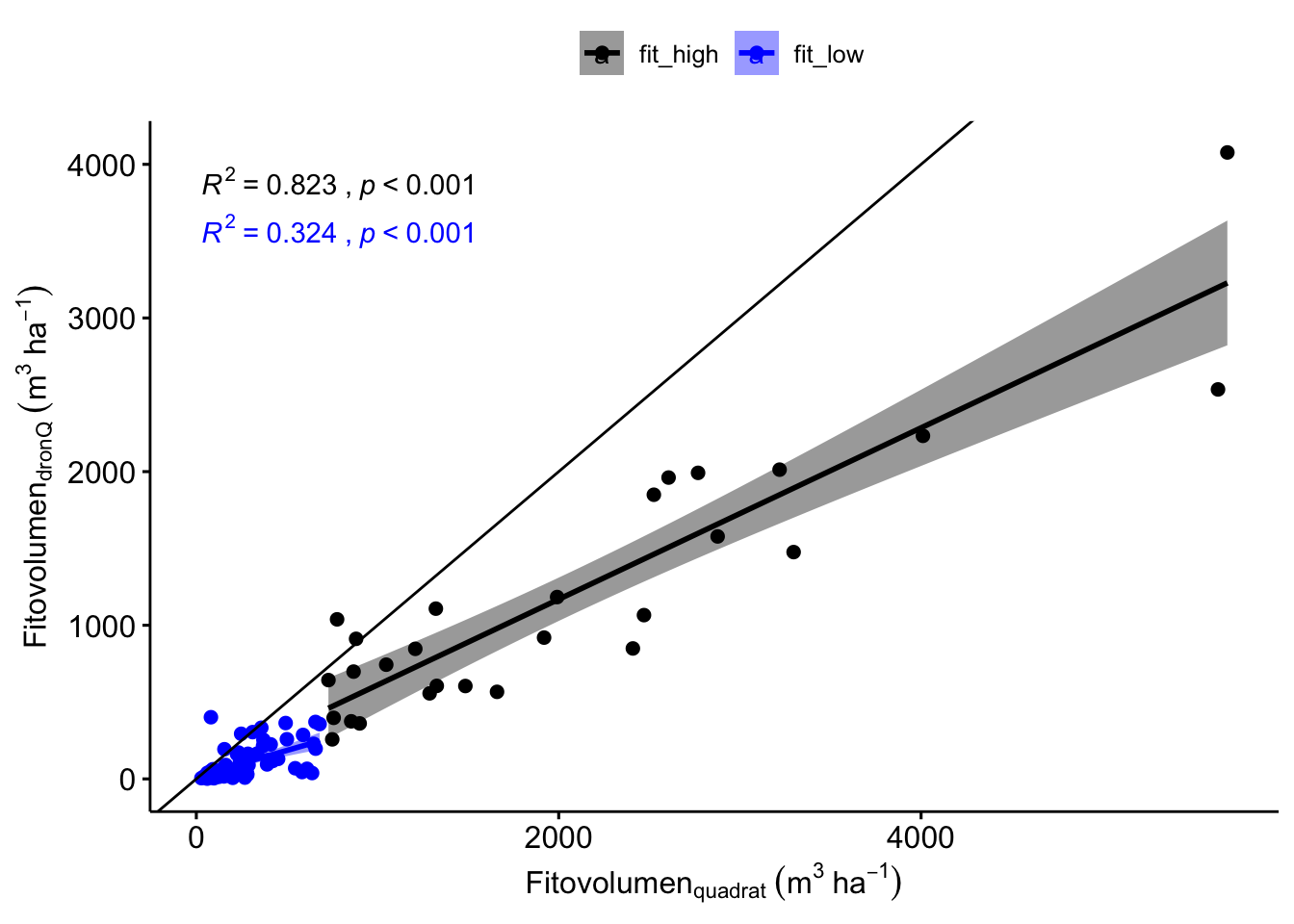
Figure 3.14: Correlación entre los valores de fitovolumen estimados por dronQ y Quadrat, considerando dos grupos (Fitovolumen inferiores (azul) y superiores (negro) a 681 % (en campo).
| clase | rmse | min | max | rmsen.minmax |
|---|---|---|---|---|
| fit_high | 1096.73 | 729 | 5690 | 22.11 |
| fit_low | 215.68 | 28 | 680 | 33.07 |
3.4 Fitovolumen dronT ~ LI
3.4.1 Summary values
| metodo | mean | sd | se | cv | median | min | max | n |
|---|---|---|---|---|---|---|---|---|
| dronT | 450.63 | 141.74 | 40.92 | 31.45 | 408.39 | 286.29 | 681.28 | 12 |
| line_intercept | 531.04 | 274.90 | 79.36 | 51.77 | 543.13 | 84.27 | 1016.86 | 12 |
3.4.2 Comparación de métodos
Los datos son normales (W = 0.97; p=0.7087); pero no se cumple la homocedasticidad (Bartlett’s K-squared = 4.31; p=0.0378). Aplicamos una comparación no paramétrica (Wilcoxon Test), y observamos que no existen diferencias significativas entre los valores de fitovolumen estimados en el line intercept frente a los estimados por el dronT (parcela) (Tabla 3.17; Figura 3.15).
| statistic | p.value | method | alternative |
|---|---|---|---|
| 59 | 0.478 | Wilcoxon rank sum exact test | two.sided |
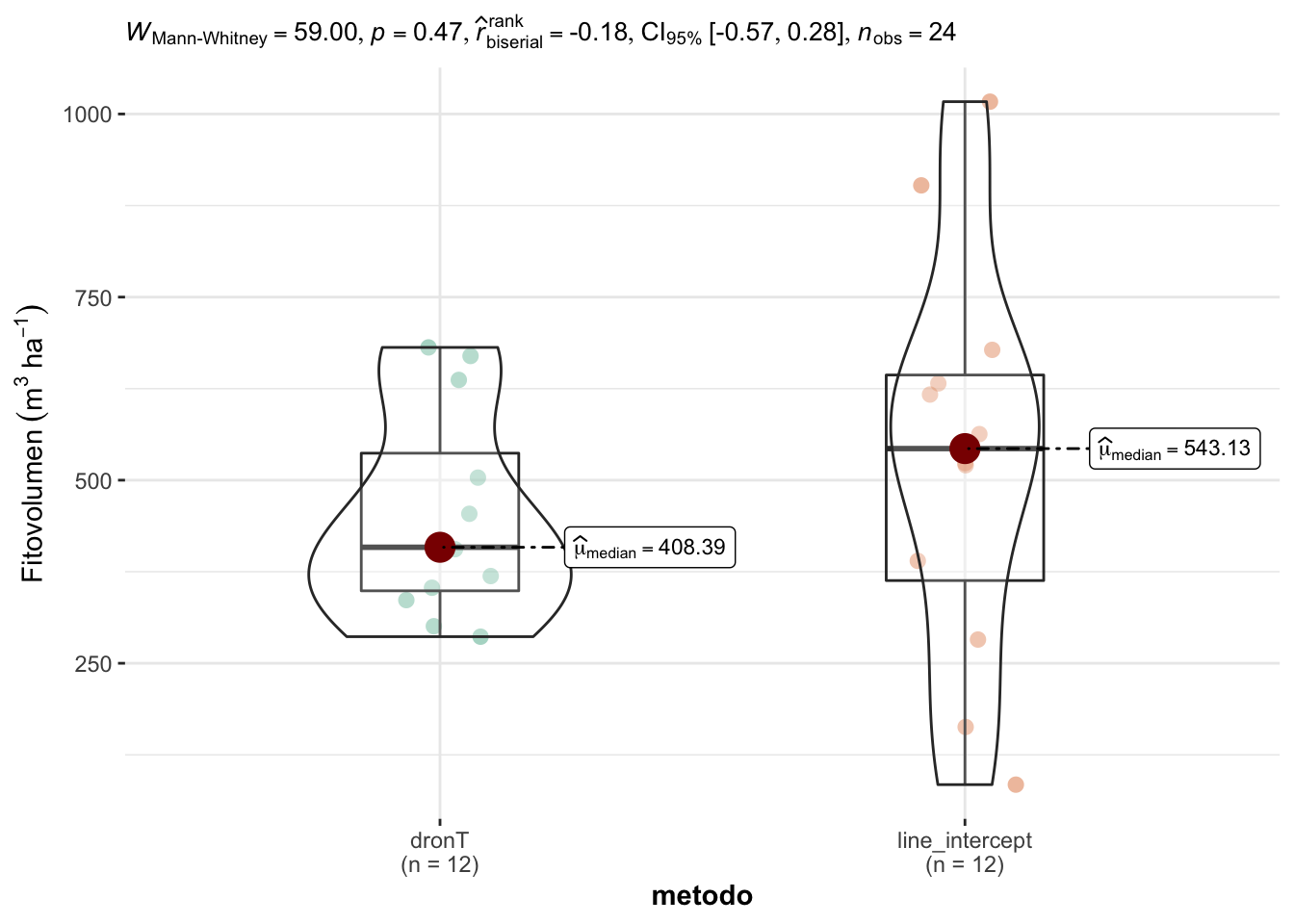
Figure 3.15: Comparación de los valores de fitovolumen entre Dron Parcela y Line Intercept.
3.4.3 Correlación
No existe una buena correlación entre los valores de fitovolumen ofrecidos por line intercept y los datos por el dron en la parcela de 500 m^2 (Figura 3.16).
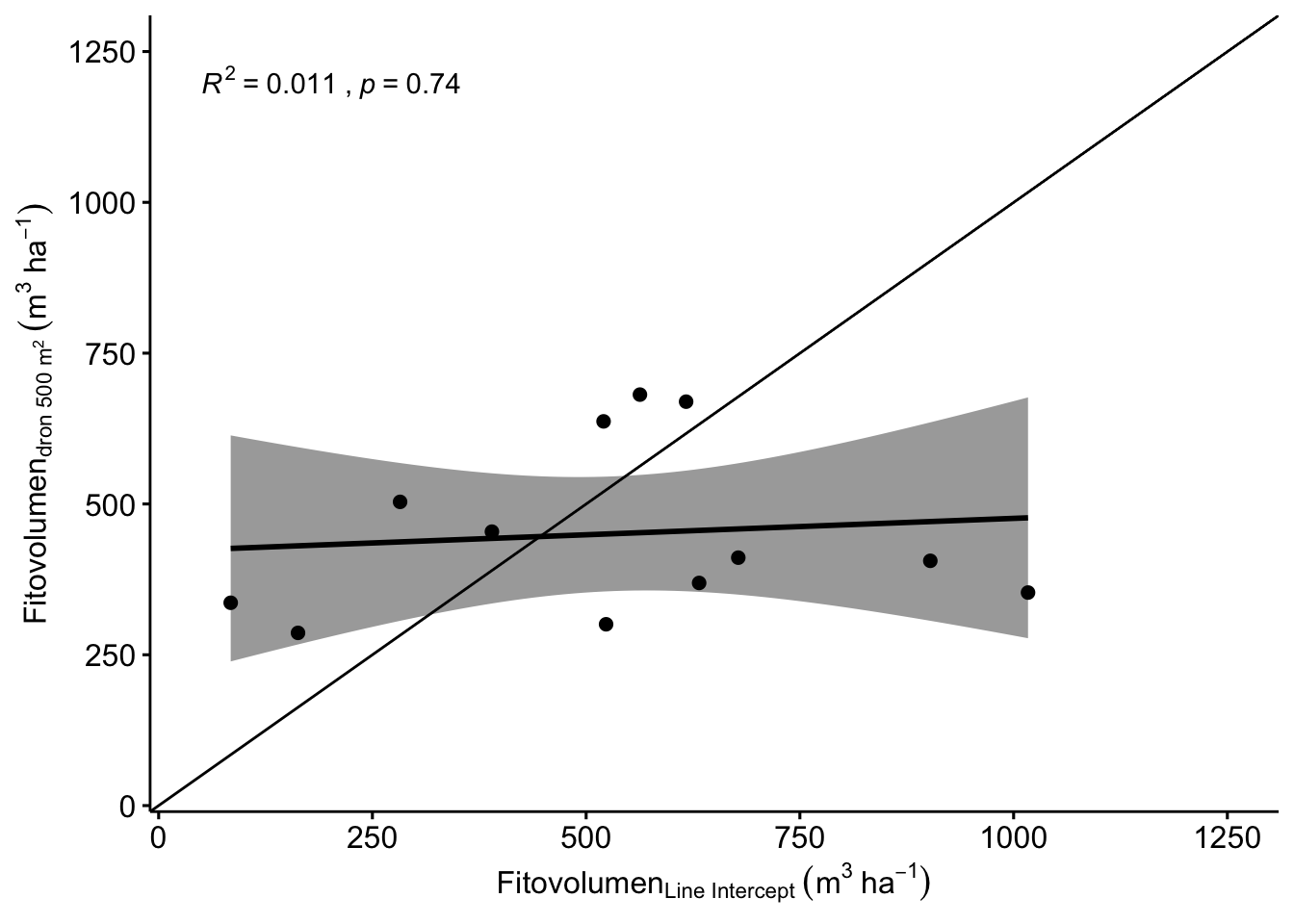
Figure 3.16: Correlación entre los valores de fitovolumen estimados por dronParcela con respecto a Line Intercept
R version 4.0.2 (2020-06-22)
Platform: x86_64-apple-darwin17.0 (64-bit)
Running under: macOS Catalina 10.15.3
Matrix products: default
BLAS: /Library/Frameworks/R.framework/Versions/4.0/Resources/lib/libRblas.dylib
LAPACK: /Library/Frameworks/R.framework/Versions/4.0/Resources/lib/libRlapack.dylib
locale:
[1] en_US.UTF-8/en_US.UTF-8/en_US.UTF-8/C/en_US.UTF-8/en_US.UTF-8
attached base packages:
[1] stats4 grid stats graphics grDevices utils datasets
[8] methods base
other attached packages:
[1] multcompView_0.1-8 ggparty_1.0.0
[3] partykit_1.2-15 libcoin_1.0-7
[5] party_1.3-9 strucchange_1.5-2
[7] sandwich_3.0-0 zoo_1.8-8
[9] modeltools_0.2-23 mvtnorm_1.1-1
[11] rpart_4.1-15 statsExpressions_1.3.1
[13] ggsignif_0.6.3 pairwiseComparisons_3.1.3
[15] ggtext_0.1.1 PMCMRplus_1.9.3
[17] PMCMR_4.3 statmod_1.4.36
[19] tweedie_2.3.3 report_0.5.1
[21] kableExtra_1.3.1 cvequality_0.2.0
[23] performance_0.8.0 ggdist_3.0.1
[25] Metrics_0.1.4 ggstatsplot_0.9.1
[27] colorspace_2.0-2 ggpubr_0.4.0
[29] ggforce_0.3.2 ggdark_0.2.1
[31] janitor_2.1.0 here_1.0.1
[33] forcats_0.5.1 stringr_1.4.0
[35] dplyr_1.0.6 purrr_0.3.4
[37] readr_1.4.0 tidyr_1.1.3
[39] tibble_3.1.2 ggplot2_3.3.5
[41] tidyverse_1.3.1 workflowr_1.7.0
loaded via a namespace (and not attached):
[1] utf8_1.1.4 tidyselect_1.1.1 gmp_0.6-2
[4] munsell_0.5.0 effectsize_0.6.0.1 codetools_0.2-18
[7] withr_2.4.1 highr_0.8 knitr_1.31
[10] rstudioapi_0.13 ipmisc_5.0.2 labeling_0.4.2
[13] emmeans_1.5.4 git2r_0.28.0 polyclip_1.10-0
[16] farver_2.1.0 datawizard_0.4.0 rprojroot_2.0.2
[19] coda_0.19-4 vctrs_0.3.8 generics_0.1.0
[22] TH.data_1.0-10 xfun_0.23 BWStest_0.2.2
[25] R6_2.5.1 BayesFactor_0.9.12-4.3 cachem_1.0.4
[28] reshape_0.8.8 assertthat_0.2.1 promises_1.2.0.1
[31] scales_1.1.1.9000 multcomp_1.4-16 gtable_0.3.0
[34] processx_3.5.1 rlang_0.4.12 MatrixModels_0.4-1
[37] zeallot_0.1.0 splines_4.0.2 rstatix_0.6.0
[40] prismatic_1.0.0 checkmate_2.0.0 broom_0.7.9
[43] yaml_2.2.1 abind_1.4-5 modelr_0.1.8
[46] backports_1.2.1 httpuv_1.5.5 gridtext_0.1.4
[49] inum_1.0-3 tools_4.0.2 bookdown_0.21.6
[52] ellipsis_0.3.2 jquerylib_0.1.3 WRS2_1.1-3
[55] Rcpp_1.0.7 plyr_1.8.6 ps_1.5.0
[58] pbapply_1.4-3 correlation_0.8.0 ggrepel_0.9.1
[61] haven_2.3.1 fs_1.5.0 magrittr_2.0.1
[64] data.table_1.14.0 openxlsx_4.2.3 reprex_2.0.0
[67] whisker_0.4 matrixStats_0.58.0 hms_1.0.0
[70] patchwork_1.1.1 evaluate_0.14 xtable_1.8-4
[73] rio_0.5.16 readxl_1.3.1 rstantools_2.1.1
[76] compiler_4.0.2 crayon_1.4.1 htmltools_0.5.2
[79] mgcv_1.8-33 mc2d_0.1-18 later_1.1.0.1
[82] Formula_1.2-4 lubridate_1.7.10 DBI_1.1.1
[85] SuppDists_1.1-9.5 kSamples_1.2-9 tweenr_1.0.1
[88] dbplyr_2.1.1 MASS_7.3-53 boot_1.3-26
[91] Matrix_1.3-2 car_3.0-10 cli_2.5.0
[94] parallel_4.0.2 insight_0.17.0 pkgconfig_2.0.3
[97] getPass_0.2-2 coin_1.4-0 foreign_0.8-81
[100] xml2_1.3.2 paletteer_1.3.0 bslib_0.2.4
[103] webshot_0.5.2 estimability_1.3 rvest_1.0.0
[106] snakecase_0.11.0 distributional_0.3.0 callr_3.7.0
[109] digest_0.6.27 parameters_0.17.0 rmarkdown_2.8
[112] cellranger_1.1.0 curl_4.3 gtools_3.8.2
[115] nlme_3.1-152 lifecycle_1.0.1 jsonlite_1.7.2
[118] carData_3.0-4 viridisLite_0.4.0 fansi_0.4.2
[121] pillar_1.6.1 lattice_0.20-41 fastmap_1.1.0
[124] httr_1.4.2 survival_3.2-7 glue_1.4.2
[127] bayestestR_0.11.5 zip_2.1.1 stringi_1.7.4
[130] sass_0.3.1 rematch2_2.1.2 memoise_2.0.0
[133] Rmpfr_0.8-2